Aspects de la signalisation cellulaire Droit d'auteur © par Charlotte de Araujo est sous licence License Creative Commons Attribution - Pas d’utilisation commerciale 4.0 International, sauf indication contraire.
Aspects de la signalisation cellulaire Droit d'auteur © par Charlotte de Araujo est sous licence License Creative Commons Attribution - Pas d’utilisation commerciale 4.0 International, sauf indication contraire.
1
Cette ressource fait le survol de la signalisation cellulaire et expose le lectorat au langage des voies cellulaires. Le style mixte enquête-solution a été choisi puisqu’il permet de diviser les voies de signalisation biochimiques en modules plus faciles à gérer et d’offrir un contenu accessible.
1
Les protéines subissent plusieurs modifications post-traductionnelles qui en augmentent les fonctions et en modifient l’interactome. La phosphorylation est l’un des mécanismes de régulation les plus courants et les plus critiques pour les protéines. Cette unité vise à donner un aperçu des principaux groupes d’enzymes intervenant dans la phosphorylation et la déphosphorylation des protéines.
À la fin de ce chapitre, vous pourrez :
Les kinases sont un terme générique désignant les enzymes qui interviennent dans le transfert d’un groupement phosphate d’une molécule (substrat) à une autre. Dans les eucaryotes, les protéines kinases conventionnelles introduisent des groupements phosphate sur trois principaux acides aminés : la sérine (S), la thréonine (Thr) ou la tyrosine (Tyr).
i. Tyrosine kinases
ii. Sérine/thréonine kinases
Le nom de la kinase correspond aux acides aminés spécifiques qui sont phosphorylés. Il existe également des kinases à double spécificité qui phosphorylent les résidus de S, Thr et Tyr. En principe, la phosphorylation des protéines sérine et thréonine est associée à d’importants changements dans la conformation des protéines, tandis que la phosphorylation des protéines tyrosine est associée à la modification de la localisation cellulaire d’une protéine. Le génome humain compte environ 518 protéines identifiées comme kinases (Manning et coll., 2002).
a. Hydroxyle (OH)
b. Thiol (SH)
c. Oxyde de diéthyle (-O-)
d. Alcène (C=C)
Lorsque l’on compare les structures de S, Thr et Tyr, on remarque que tous ces acides aminés possèdent un groupe hydroxyle dans leur groupe de chaîne latérale (figure 1.1). Un groupement phosphate chargé négativement peut être conjugué à ces acides aminés, produisant respectivement la phosphosérine, la phosphothréonine ou la phosphotyrosine.
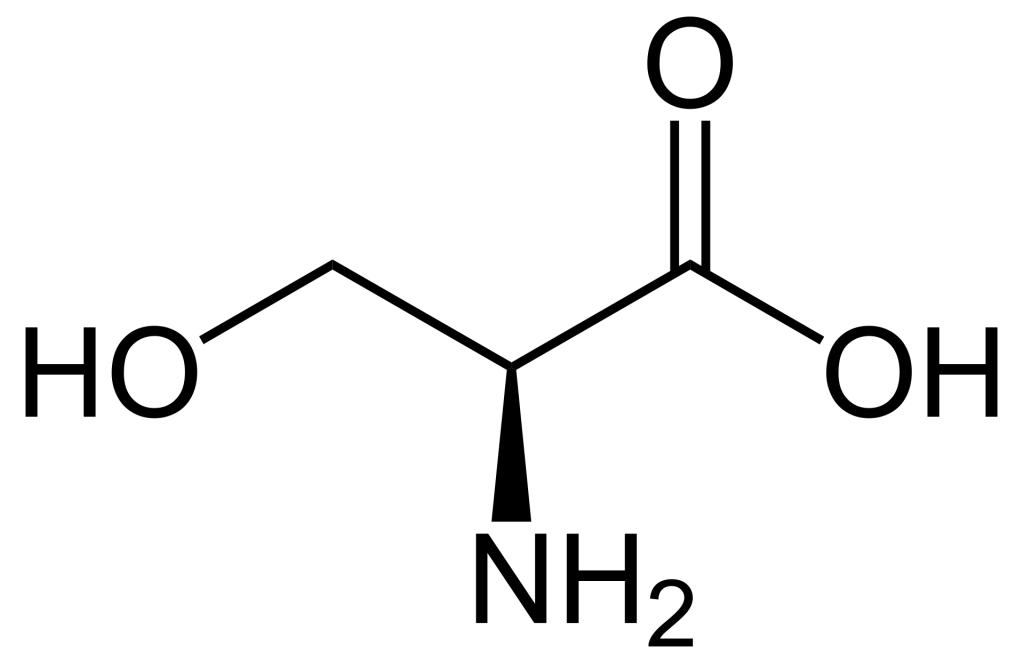
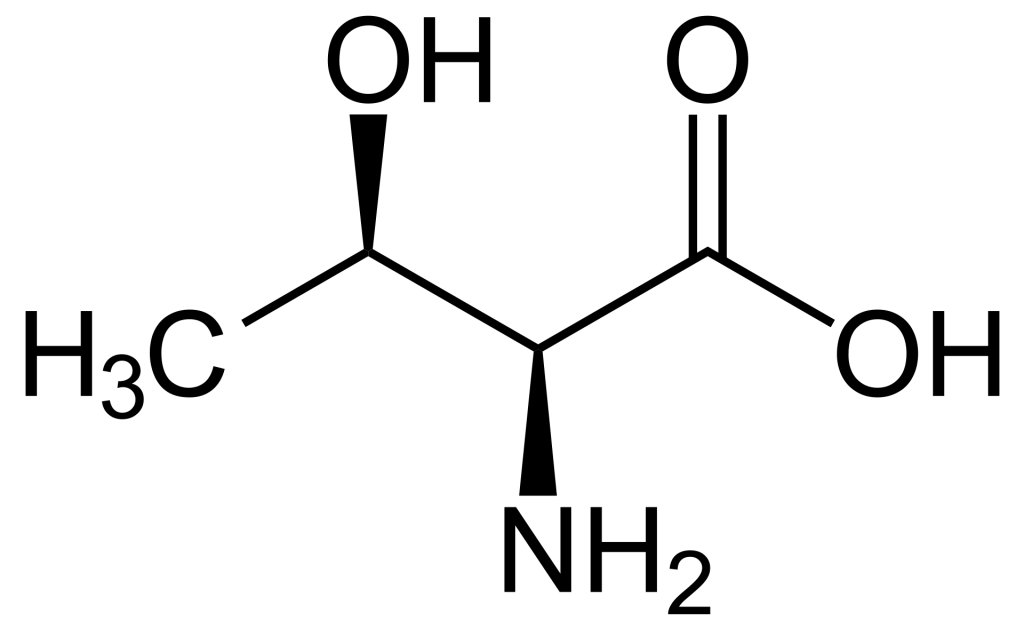
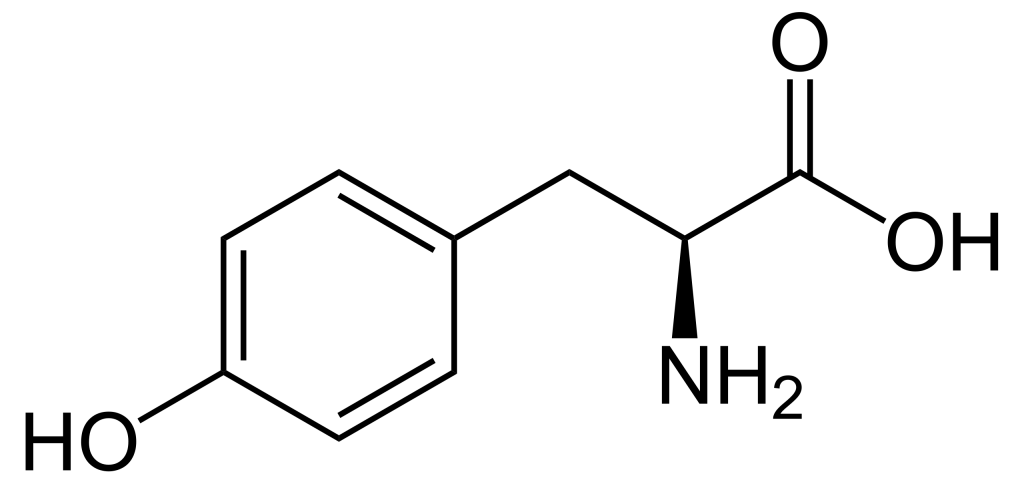
a. Elle augmente l’activité catalytique de la protéine.
b. Elle diminue l’activité catalytique de la protéine.
c. Cela dépend du scénario.
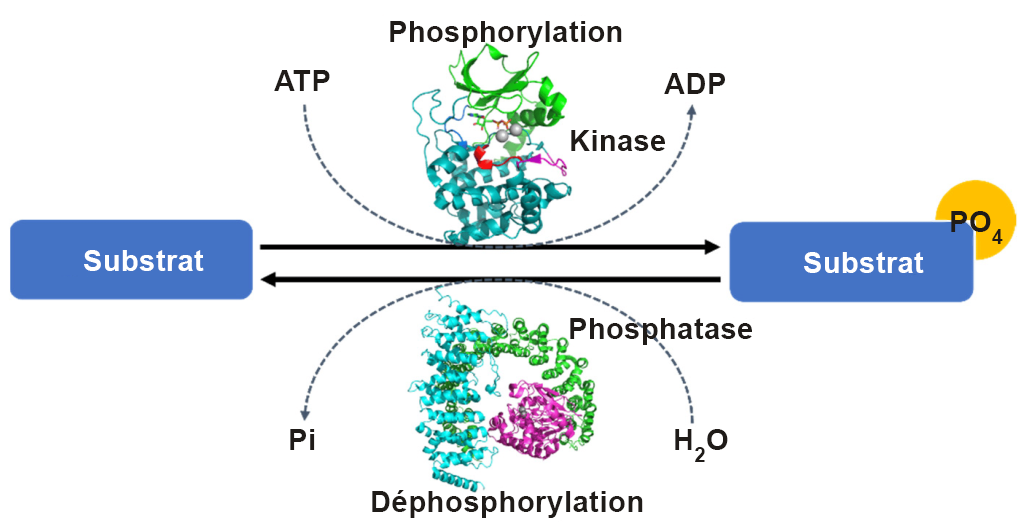
La phosphorylation d’une protéine (ajout d’un groupement phosphate) peut augmenter ou diminuer l’activité catalytique d’une protéine selon les changements structurels qui en résultent et qui ont une incidence sur la conformation de la protéine (figure 1.2). Le groupement phosphate introduit une charge négative divalente sur le site de la protéine (distincte de tout acide aminé naturellement présent) qui peut modifier radicalement les propriétés physico-chimiques de la protéine.
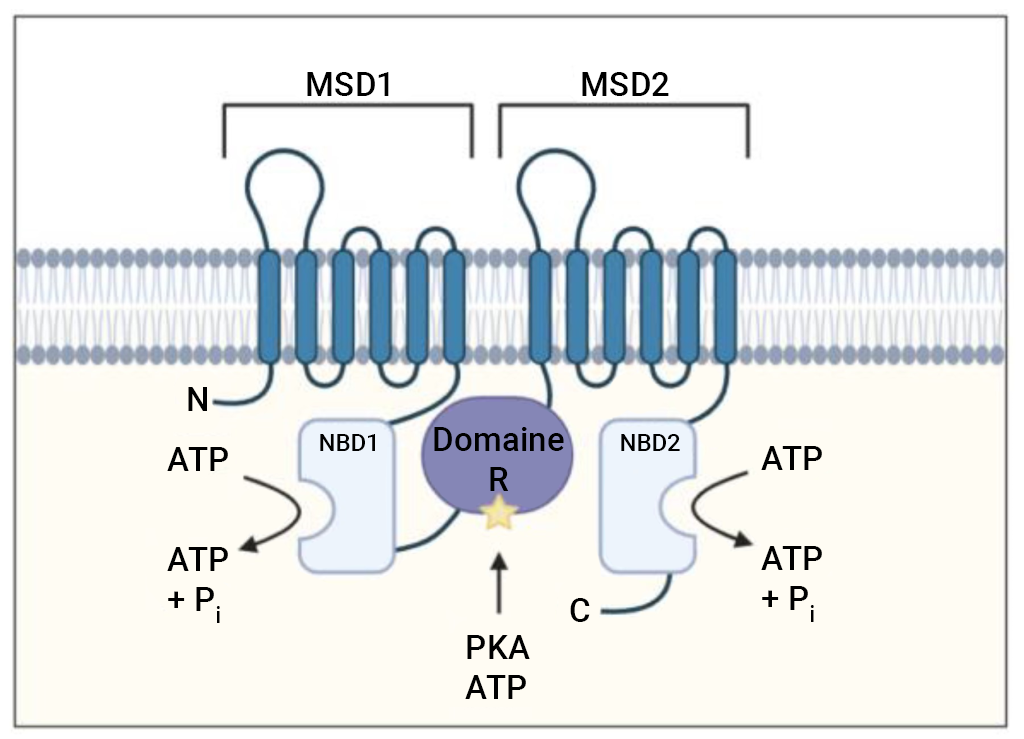
Par exemple, la phosphorylation d’une protéine de type canal ionique chlorure appelée CFTR (régulateur de la perméabilité transmembranaire de la fibrose kystique) se produit sur une boucle spécifique (appelée région R) de la protéine, qui bloque normalement le flux d’ions chlorure à travers le canal (figure 1.3). La perturbation électrostatique résultant de la phosphorylation de la boucle lui permet d’interagir avec d’autres régions de la protéine, ce qui l’empêche de bloquer stériquement l’accès aux pores.
De plus, un sous-ensemble de protéines est également sujet à la phosphorylation sur plusieurs sites, où il existe des grappes de S ou de Thr pouvant être phosphorylées. L’augmentation des niveaux de phosphorylation peut entraîner un effet plus prononcé. Par exemple, la région R du CFTR contient au moins dix sites de phosphorylation, et une phosphorylation progressive augmente l’activité de régulation.
En biologie, il existe plusieurs catégories se terminant par « ome ». Le suffixe « ome » désigne généralement un sous-ensemble de biomolécules provenant d’une cellule ou d’un organisme. Il peut s’agir de sous-ensembles définis de façon plus large, comme le génome (sous-ensemble de gènes au sein d’une cellule) ou le protéome (sous-ensemble de protéines au sein d’une cellule). Il existe également des termes plus spécialisés, comme le « kinome », qui englobe toutes les kinases d’un organisme. Le kinome humain est souvent représenté sous la forme d’un « arbre du kinome », qui est une représentation visuelle phylogénétique des relations évolutives entre les 518 kinases différentes (Manning et coll., 2002).
La sérine, la thréonine et la tyrosine sont des résidus couramment présents dans les protéines. Toutefois, ces résidus ne sont pas tous marqués par un groupement phosphate sous l’action d’une kinase. Les kinases reconnaissent une séquence spécifique contenant la S, Thr et Tyr, appelée « séquence consensus ». Cette séquence peptidique fournit un motif de reconnaissance permettant la liaison de la kinase, ce qui permet des interactions spécifiques avec le site actif de la kinase afin d’insérer les résidus de S, Thr et Tyr appropriés dans la pochette. Par exemple, la séquence de reconnaissance de la protéine kinase dépendante (PKA) de l’AMPc est R-R/K-x-S-ϕ (où X est un acide aminé quelconque et ϕ un résidu hydrophobe) (Kemp et coll., 1977).
a. Les enzymes qui catalysent l’hydrolyse des groupes phosphoryles sur les substrats de protéine.
b. Les enzymes qui catalysent l’ajout de groupes phosphoryles sur les substrats de protéine.
c. Les enzymes qui catalysent la formation de liaisons disulfures.
L’un des avantages de la phosphorylation en tant qu’événement post-traductionnel d’une protéine est que le groupement phosphate peut être éliminé de manière réversible par des étapes de déphosphorylation (figure 1.2). Cela crée un mécanisme de régulation transitoire, en agissant efficacement comme une séquence activatrice pour activer ou désactiver une protéine. Les protéines phosphatases sont des enzymes qui éliminent les groupements phosphate présents sur les acides aminés S, Thr ou Tyr, ce qui entraîne un retour à la chaîne latérale d’origine de la protéine avec un groupe hydroxyle (-OH). Un groupe d’orthophosphate (Pi) est ensuite libéré.
Comme pour les kinases, il existe deux classes communes de phosphatases qui participent aux voies de signalisation cellulaires :
i. Protéines S/Thr phosphatases
ii. Protéines Tyr phosphatases
Les protéines tyrosine phosphatases sont des enzymes qui catalysent l’élimination des groupements phosphate des résidus Tyr présents dans les substrats de protéine. Les protéines tyrosine phosphatases peuvent être classées en deux catégories : les protéines de type récepteur et les protéines non transmembranaires. Les protéines tyrosine phosphatases de type récepteur seront abordées dans le module sur les récepteurs couplés aux enzymes. Il existe environ 226 protéines phosphatases (Liu et Chance, 2014).
Le domaine d’homologie avec Src-2 (SH2) contenant la protéine tyrosine phosphatase 2 ou SHP2 est un exemple de membre d’une protéine phosphatase non transmembranaire, exprimée dans plusieurs tissus. La SHP2 est un exemple particulier de phosphatase cytosolique, composée de différents domaines de protéines, dont deux domaines SH2 en tandem et un domaine de phosphatase catalytique (Qu, 2000). La SHP2 agit en déphosphorylant plusieurs protéines, ce qui a généralement un rôle négatif dans le blocage des voies de signalisation.
En l’absence d’une protéine cible, le domaine de phosphatase interagit avec la région N-terminale du domaine SH2 de la SHP2. Cette interaction maintient la protéine dans une conformation fermée et empêche l’accès des substrats au site de la phosphatase. Par conséquent, la SHP2 est incapable d’effectuer des réactions d’hydrolyse du phosphate.
En présence d’une cible (p. ex., une protéine contenant de la tyrosine phosphorylée), la cible phosphorylée liera le domaine SH2 de la SHP2. Cet événement de liaison déclenche un changement conformationnel, modifiant les interactions entre le domaine SH2 et le domaine catalytique. Le site actif devient plus accessible, ce qui permet la liaison de la protéine au domaine catalytique de la phosphatase et l’élimination ultime du groupement phosphate de la protéine cible. Les produits sont libérés pour réinitialiser le cycle catalytique.
Au sein du groupe des protéines tyrosine phosphatases, il existe une autre sous-famille, appelée « phosphatases à double spécificité », qui sont des phosphatases hydrolysant les groupements phosphate, principalement à partir des résidus de Tyr. Cependant, ils possèdent également une activité phosphatase des protéines S et Thr.
Harwood, K. H., McQuade, R. M., Jarnicki, A., et Schneider-Futschik, E. K. (2021). Anti-Inflammatory Influences of Cystic Fibrosis Transmembrane Conductance Regulator Drugs on Lung Inflammation in Cystic Fibrosis. International Journal of Molecular Sciences, 22(14). https://doi.org/10.3390/ijms22147606
Kemp, B. E., Graves, D. J., Benjamini, E., et Krebs, E. G. (1977). Role of multiple basic residues in determining the substrate specificity of cyclic AMP-dependent protein kinase. The Journal of Biological Chemistry, 252(14), 4888-4894. https://www.jbc.org/article/S0021-9258(17)40137-2/pdf
Liu, Y., et Chance, M. R. (2014). Integrating phosphoproteomics in systems biology. Computational and Structural Biotechnology Journal, 10(17), 90-97. https://doi.org/10.1016%2Fj.csbj.2014.07.003
Manning, G., Whyte, D. B., Martinez, R., Hunter, T., et Sudarsanam, S. (2002). The Protein Kinase Complement of the Human Genome. Science, 298(5600), 1912-1934. https://doi.org/10.1126/science.1075762
Qu, C. K. (2000). The SHP—2 tyrosine phosphatase:Signaling mechanisms and biological functions. Cell Research, 10(4), 279-288. https://doi.org/10.1038/sj.cr.7290055
Seok, S.-H. (2021). Structural Insights into Protein Regulation by Phosphorylation and Substrate Recognition of Protein Kinases/Phosphatases. Life, 11(9). https://doi.org/10.3390/life11090957
2
Les processus biochimiques fondamentaux dans le corps reposent sur des cascades de signalisation enzymatique élaborées et très complexes. Ces voies de signalisation peuvent employer des récepteurs situés à la surface cellulaire (p. ex., récepteurs couplés à des enzymes, récepteurs couplés aux protéines G) ou récepteurs nucléaires. Cette unité examinera les propriétés structurelles et fonctionnelles de divers récepteurs couplés à des enzymes.
Le terme récepteurs couplés à des enzymes peut être considéré comme réunissant deux composantes discrètes : les récepteurs et les enzymes. Ces protéines sont des récepteurs qui ont aussi une activité enzymatique – la capacité de catalyser une réaction. Les récepteurs sont des protéines transmembranaires qui interagissent avec un signal ou un ligand. Cette interaction se produit du côté extracellulaire de la cellule et entraîne un changement conformationnel de la protéine menant à une activité enzymatique du côté intracellulaire de la cellule. De cette façon, les récepteurs couplés à des enzymes activent les processus de signalisation cellulaire.
Il existe six grandes catégories de récepteurs couplés à des enzymes (Alberts, 2002).
I. Récepteur tyrosine kinase (RTK)
II. Récepteur associé à la tyrosine kinase
III. Récepteur sérine-thréonine kinase
IV. Récepteur associé à l’histidine kinase
V. Récepteur guanylyl cyclase
VI. Tyrosine phosphatase de type récepteur
À la fin de ce chapitre, vous pourrez :
Il existe 58 RTK connus dans le génome humain et ils réagissent à différents signaux extracellulaires (Robinson et coll., 2000). Cependant, tous les RTK possèdent des domaines structurels communs : un domaine de liaison extracellulaire du ligand, une hélice transmembranaire, une région juxtamembranaire et un domaine de la tyrosine kinase (TK).
Les RTK sont des protéines intégrales qui traversent la membrane cellulaire (par le biais du domaine transmembranaire, qui consiste en une seule hélice). La région N-terminale du récepteur est située dans la région extracellulaire, tandis que la partie C-terminale est située dans le cytoplasme.
La région N-terminale du RTK contient le site de liaison extracellulaire du ligand, c’est-à-dire l’emplacement où un signal externe interagira avec la protéine. Cette région extracellulaire varie grandement entre les différents RTK et peut inclure des régions riches en cystéine, des segments riches en leucine ou des motifs semblables à l’immunoglobuline. Cela permet de maintenir la diversité pour la reconnaissance de différents signaux biochimiques externes (Lemmon et Schlessinger, 2010).
La région C-terminale existe à l’intérieur de la cellule (le cytosol) et comprend le domaine de la tyrosine kinase et la région juxtamembranaire conservés. Ce domaine kinase exécute l’activité de phosphorylation du RTK, tandis que la région juxtamembranaire est très souple et importante pour les rôles de régulation, ce qui entraîne souvent une auto-inhibition à la suite de contacts avec le domaine kinase.
a. Sérine
b. Thréonine
c. Tyrosine
d. a et b
Les RTK phosphorylent les résidus de tyrosine sur les substrats de la protéine.
a. α
b. β
c. γ
d. α et β
e. β et γ
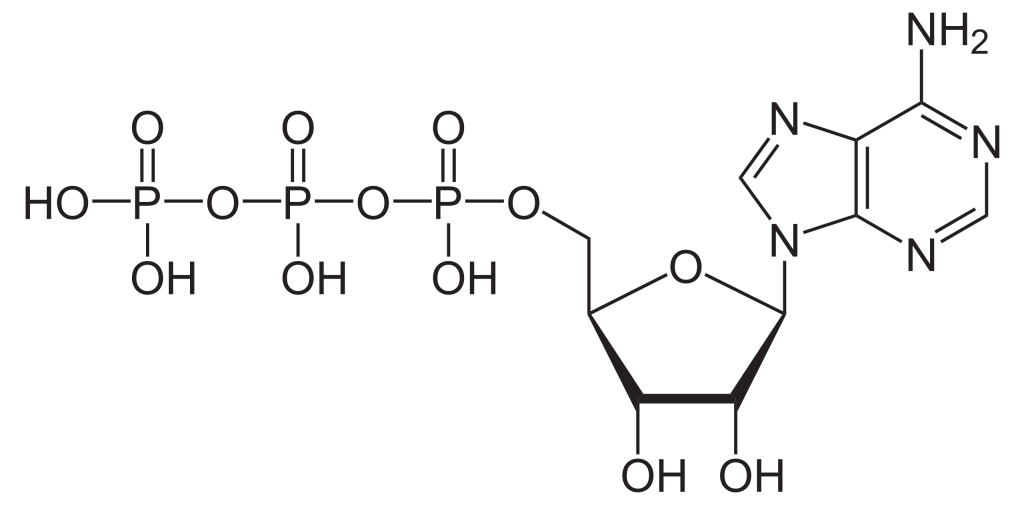
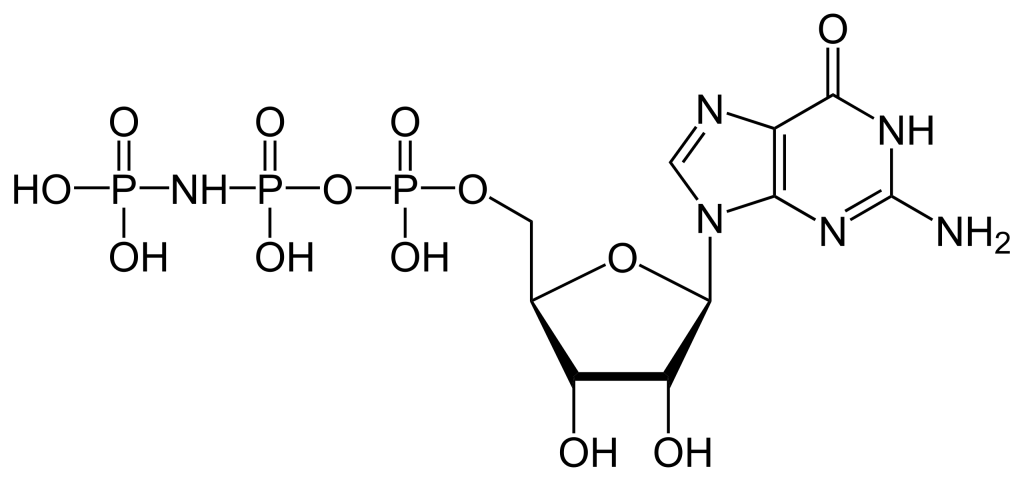
Pour construire le nucléotide, l’ATP, trois composants essentiels sont nécessaires : le ribose à cinq atomes de carbone, une base azotée (adénine) et trois groupements phosphate étiquetés, α, β et γ (figure 2.1). Le phosphate α est placé le plus près du groupe de sucre, tandis que le phosphate γ est le plus éloigné. Les RTK transfèrent un groupement phosphate de l’ATP, ajoutant un groupement phosphate à la tyrosine sur les substrats de la protéine. Le groupement phosphate γ est transféré à l’étape de phosphorylation vers un substrat de protéine. Il s’agit d’une réaction très exergonique.
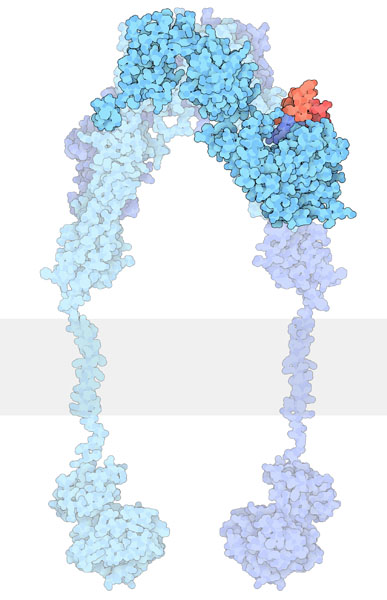
À l’« état inactif », les RTK existent généralement sous la forme d’une seule protéine appelée monomère. Toutefois, la forme « active » nécessite que deux protéines RTK se trouvent à proximité, ce que l’on appelle un dimère (figure 2.2). Il y a toutefois des exceptions. Par exemple, le récepteur de l’insuline (IR) existe également comme dimère sous la forme inactive (figure 2.3) en raison d’un pont disulfure à liaison cystéine entre les espèces monomériques.
Pour ce mécanisme, nous supposerons que les RTK se présentent initialement sous forme monomérique inactive. Lorsqu’un ligand approprié se lie au récepteur au niveau du domaine de liaison du ligand extracellulaire, cela entraîne un changement conformationnel qui convertit le RTK inactif à la forme active et facilite le regroupement de deux RTK monomériques. Ces deux monomères sont placés à proximité l’un de l’autre, ce qui entraîne la formation d’un dimère.
Rappelez-vous que le domaine TK se trouve dans la partie cytosolique du récepteur. Une fois que le dimère s’est formé, les domaines TK sont positionnés de manière à exécuter la « trans-autophosphorylation ». C’est à ce moment qu’un domaine TK d’un monomère phosphoryle des résidus de tyrosine ciblés situés sur le monomère opposé. Cette « trans-phosphorylation » convertit le domaine kinase en domaine kinase actif, et le RTK inactif est converti en RTK actif.
Oui, la liaison du ligand (p. ex., facteur de croissance épidermique, EGF) au domaine de liaison du ligand du récepteur (p. ex., récepteur du facteur de croissance épidermique, récepteur EGF) provoque des changements conformationnels, activant finalement l’activité enzymatique du RTK (figure 2.2). De cette façon, les signaux externes à la cellule sont transmis à l’intérieur de celle-ci, sans que la molécule ou l’hormone traverse la membrane.
Dans ce RTK muté, on suppose que les domaines transmembranaire et cytoplasmique sont intacts sur le plan fonctionnel et structurel. Cependant, comme le domaine de liaison du ligand est absent, il n’y a pas de réponse de régulation et le RTK n’est plus sensible au ligand qui y est associé. Dans ce cas, le récepteur sera dans un état « actif » perpétuel ou constitutivement actif. Il y a une conséquence négative à cela, à savoir qu’un récepteur indépendant du ligand peut entraîner une hyperphosphorylation et une hyperactivation des voies RTK en aval. En fait, il a été démontré que 30 % des cancers humains présentaient des RTK hyperactivés ou surexprimés (Ségaliny et coll., 2015).
a. Elle inhiberait l’activité du R-EGF.
b. Elle activerait le R-EGF.
Comme nous l’avons déjà mentionné, la SHP-2 est un phosphatase. À proximité du R-EGF, elle éliminerait probablement le groupement du R-EGF (Lemmon et Schlessinger, 2010), ce qui réduirait l’activité de ce RTK.
Après la trans-autophosphorylation, les RTK possèdent des résidus de phospho-tyrosine. Ces résidus fonctionnent comme des sites pour permettre la liaison et le recrutement de protéines de signalisation précises. Les régions du RTK qui contiennent les résidus de phospho-tyrosine sont également appelées sites d’interaction ou sites de liaison. Les protéines de signalisation qui se lient à cette région possèdent des domaines de liaison phospho-tyrosine.
a. Région extracellulaire
b. Partie transmembranaire du récepteur
c. Région intracellulaire
Certains RTK, comme l’IR, existent sous forme de dimères dans la forme inactive. Comme d’autres RTK, tel que le R-EGF, le domaine de liaison du ligand se trouve dans le domaine extracellulaire (figure 2.4).
a. Sérine
b. Méthionine
c. Cystéine
d. Cystéine et méthionine
e. Cystéine, méthionine et sérine
L’IR contient des ponts disulfures formés de deux résidus de cystéine.
Comme d’autres RTK, le ligand se lie au récepteur et cette interaction entraîne un changement conformationnel qui active le domaine tyrosine kinase. Chaque monomère (en bleu) peut phosphoryler l’unité monomérique opposée (figure 2.4). Le domaine tyrosine actif exécute l’étape de l’« autophosphorylation », phosphorylant l’autre unité au niveau des résidus de tyrosine.
a. L’activité de l’IR s’amplifierait.
b. L’activité de l’IR diminuerait.
c. Aucun effet sur l’activité de l’IR.
Le dithiothréitol est un réducteur qui cible les ponts disulfures, catalysant leur réduction. On prévoit que le traitement au dithiothréitol diminuerait l’activité des récepteurs (Pike et coll., 1986).
a. Cystéine
b. Méthionine
c. Tyrosine
L’IR est un RTK, où les résidus de tyrosine sont phosphorylés.
L’IR (à l’état actif) phosphoryle d’autres cibles cellulaires, y compris un substrat courant du récepteur de l’insuline (IRS) cible.
Les protéines associées aux voies RTK possèdent souvent des domaines SH2 (région d’homologie avec la protéine Src 2) ou des domaines PTB (liaison phospho-tyrosine) pour la liaison phospho-tyrosine. À titre d’exemple, l’IRS1 se lie au récepteur phosphorylé en utilisant son domaine PTB. Mentionnons aussi la Grb2, qui lie des motifs contenant la phospho-tyrosine par l’intermédiaire d’un domaine SH2.
Le domaine SH2 est un domaine protéique qui sert à lier les résidus de phospho-tyrosine. Le domaine SH2 comporte deux sous-pochettes formées de deux hélices alpha séparées par un feuillet β central (composée de trois brins β). Le résidu de phospho-tyrosine s’insère dans une sous-pochette (appelée « pochette pY ») d’un côté du feuillet β central et les résidus de la région C-terminale de la phospho-tyrosine s’insèrent dans l’autre sous-pochette (appelée « pochette pY + 3 ») de l’autre côté du feuillet β (Bajusz et coll., 2023). Ces résidus contigus confèrent une spécificité au moyen de chaînes latérales clés d’acides aminés. Par conséquent, différents domaines SH2 reconnaîtront les protéines contenant la phospho-tyrosine dans le contexte des acides aminés avoisinants. Ce mécanisme d’action d’un domaine SH2 est souvent comparé à une fiche et à une prise à deux broches (Waksman et coll., 1993).
L’insuline (ligand) se lie au récepteur apparenté, l’IR, situé à la surface cellulaire de tissus spécifiques (figure 2.5). Il en résulte un changement conformationnel et une autophosphorylation subséquente de résidus de tyrosine particuliers au niveau de l’IR. L’IR activé phosphoryle des cibles, incluant les substrats du récepteur de l’insuline des protéines adaptatrices (p. ex., IRS1). L’IRS1 (à son état phosphorylé) sert de site d’interaction pour une kinase, le phosphatidylinositide 3-kinase (PI3K). L’enzyme PI3K catalyse la phosphorylation du phosphatidylinositol 4,5-bisphosphate (PIP2) en phosphatidylinositol-3,4,5-triphosphate (PIP3). Le PIP3 active une autre kinase, la PDK1 (protéine kinase dépendante du PIP3). Dans sa forme active, la PDK1 phosphoryle d’autres kinases, incluant la protéine kinase B (PKB, aussi connue sous le nom d’Akt1). De même, le mTORC2 phosphoryle et active également la PKB. En continuant la série d’événements de phosphorylation, la PKB phosphoryle des cibles, incluant le glycogène synthase kinase 3 (GSK3). Dans ce cas, la phosphorylation désactive l’enzyme GSK3. Dans sa forme phosphorylée, le GSK3 est incapable de désactiver la glycogénésynthétase (GS). La GS est une enzyme qui participe à la conversion du glucose en glycogène.
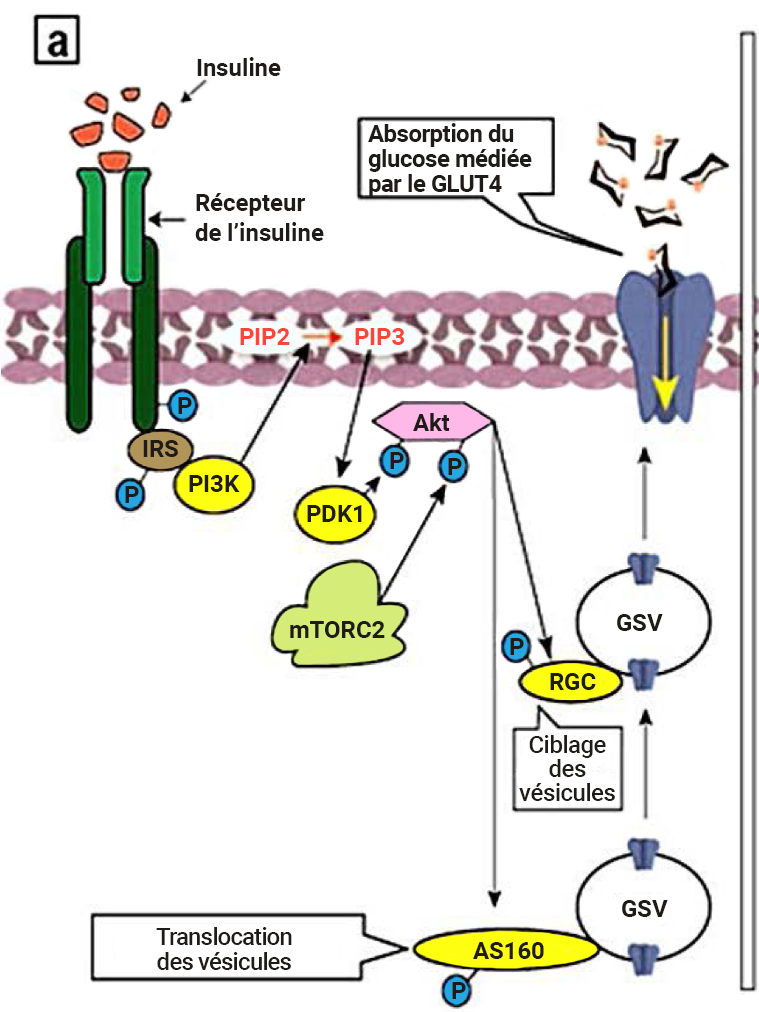
Ici, le terme mTORC2 sera analysé plus en détail. La TOR est une protéine kinase dont la forme mammalienne est appelée mTOR ou cible de la rapamycine chez les mammifères. Ensemble, deux complexes protéiques différents, mTORC1 et mTORC2, forment une mTOR. En tant que protéine kinase sérine/thréonine, la mTOR phosphoryle la sérine ou la thréonine (alors que les événements de phosphorylation originaux étaient dus à la phospho-tyrosine). Le complexe protéique mTORC2 est activé de façon classique par divers éléments allant des acides aminés aux facteurs de croissance comme l’insuline (Tato et coll., 2011; Yoon, 2017) (figure 2.5).
La PKB recrute et active également les vésicules insulinosensibles contenant du GLUT4 pour favoriser le transport du glucose. La PKB phosphoryle le GTPases, le RAB GAP AS160 et le complexe RAL-GAP (RGC) (figure 2.5) (Sayem et coll., 2018). Ces GTPases favorisent le transport des vésicules de GLUT4 vers la membrane cellulaire. Les vésicules de GLUT4 contiennent un transporteur de glucose (GLUT4) incorporé dans la membrane cellulaire. Ces protéines, qui facilitent le transport du glucose du sang vers la cellule, sont reconnues pour rendre les cellules « perméables » au glucose.
La Ras fait référence à une famille de protéines qui sont de petites GTPases monomériques. La Ras se compose d’isoformes (p. ex., H-Ras, K-Ras, N-Ras). La protéine de liaison à la GTP, Ras, a été identifiée dans les sarcomes du rat, formant ainsi la base de ce nom.
L’interrupteur d’éclairage (figure 2.6) bascule entre les options « MARCHE » et « ARRÊT », où la lumière s’allume à l’état « MARCHE » et s’éteint à l’état « ARRÊT ».
De même, lorsque la Ras est liée au nucléotide guanosine triphosphate (GTP), elle est à l’état actif.
Ras + GTP → active ou « MARCHE »
Tandis que, lorsque la Ras est liée au nucléotide guanosine diphosphate (GDP), elle est à l’état inactif.
Ras + GDP → inactive ou « ARRÊT »
Par conséquent, la Ras est analogue à un interrupteur existant sous deux formes (actif et inactif) (figure 2.6).

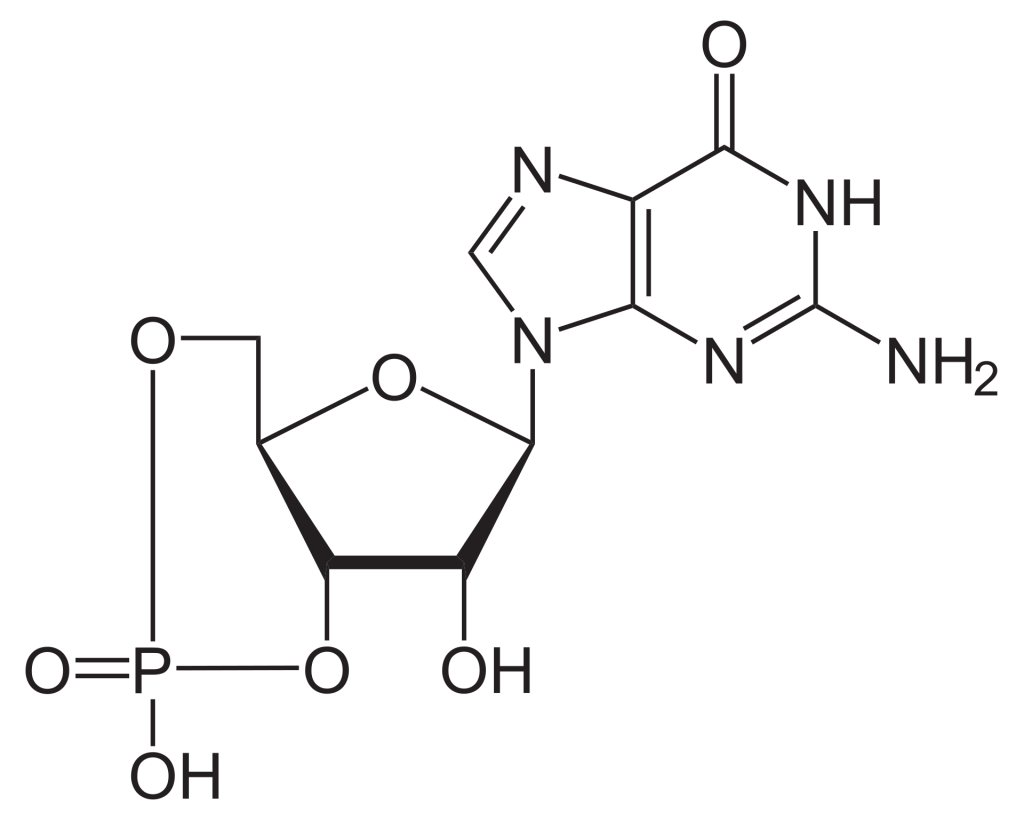
La GTP est un nucléotide composé de trois composants clés : sucre à cinq atomes de carbone (ribose), base azotée (guanine) et trois groupements phosphate. La GDP est un nucléotide formé à partir des mêmes composants, sauf qu’il possède deux groupements phosphate.
Les facteurs d’échange nucléotidiques (GEF) sont généralement des protéines qui catalysent le passage de la GDP à la GTP. Lorsque la Ras est liée à la GDP, le GEF catalyse le passage de la GDP à la GTP.
Les protéines activant la GTPase (GAP) favorisent l’hydrolyse de la GTP à la GDP. Lorsque la Ras est liée à la GTP, les GAP catalysent le passage de la GTP à la GDP. La Ras est maintenant liée à la GDP.
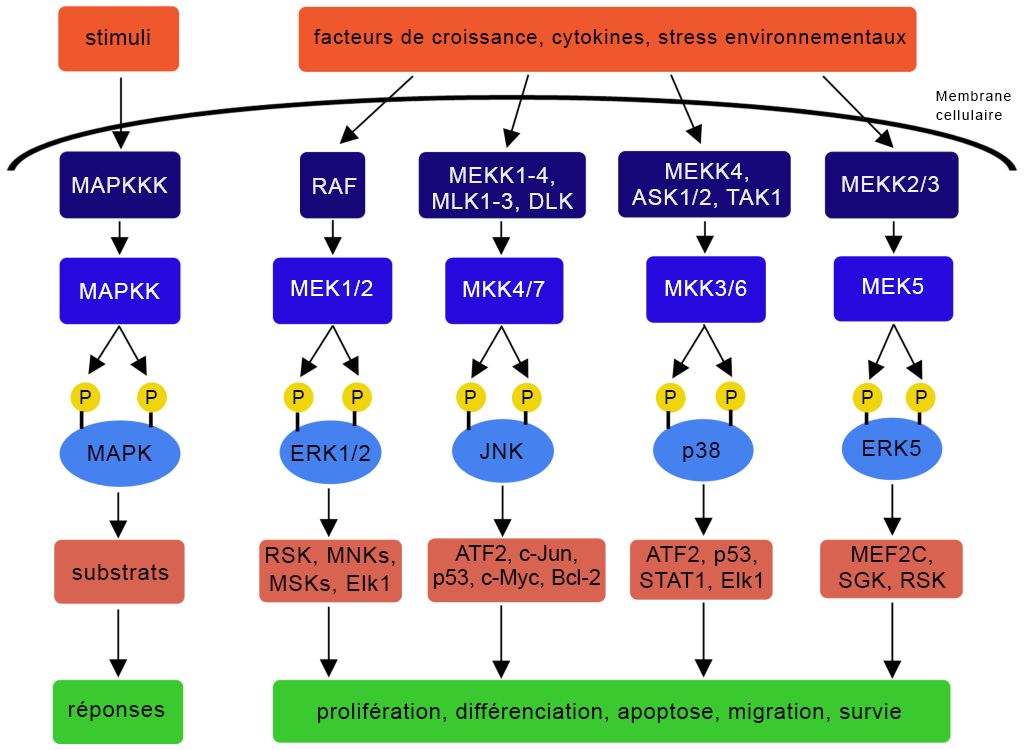
Les protéines kinases activées par des agents mitogènes (MAPK), ou kinases MAP, sont un groupe ubiquiste d’enzymes présentes dans les eucaryotes (figure 2.7). Chez les mammifères, les regroupements représentatifs suivants (i à iii) sont membres de la famille des MAPK (Morrison, 2012).
i. Protéine JNK
ii. Protéine ERK
iii. Protéines kinase activées par le mitogène p38 (p38)
a. Sérine
b. Thréonine
c. Tyrosine
d. a ou b
e. Toutes les réponses ci-dessus
Les membres de la famille des MAPK phosphorylent les résidus de sérine ou de thréonine.
Les membres de la famille des MAPKK et des MAPKKK phosphorylent la sérine ou la thréonine.
Une MAPKKK représentative, RAF1 (figure 2.7), phosphoryle des résidus précis de sérine sur la MEK. Les membres de la famille des MAPKK phosphoryle les résidus de sérine et de thréonine de la famille des MAPK.
La documentation utilise divers noms pour décrire les kinases suivantes. Associez les kinases suivantes aux autres noms utilisés pour les décrire (tableau 1).
| Kinase | Autre nom |
| MAPK | MAP kinase |
| MAPKK | MAP2K ou MAP kinase kinase |
| MAPKKK | MKKK ou M3K ou MAP3K ou MAP kinase kinase kinase |
| MAPKKKK | MAP4K ou MAP kinase kinase kinase kinase |
À l’intérieur de la voie de signalisation MAPK, il y a une série d’événements de phosphorylation dans lesquels la MAPKKK phosphoryle la MAPKK, qui, elle, phosphoryle la MAPK (figure 2.7). Un relais ou une « cascade » de réactions de phosphorylation a lieu (Meister et coll., 2013). La MAPK procédera ensuite à la phosphorylation de protéines supplémentaires, comme les facteurs de transcription.
La MEK phosphoryle les résidus de sérine et de thréonine ou de tyrosine sur le substrat de protéine (tableau 2). Par exemple, la MEK phosphoryle des résidus de thréonine et de tyrosine bien précis de la protéine ERK (Zheng et Guan, 1993).
| Composante | Rôle |
| Ras | Protéine G monomérique |
| RAF | Sérine/thréonine kinase |
| MEK | Protéine kinase à double spécificité |
| ERK | Sérine/thréonine kinase |
La voie MAPK intervient dans un éventail d’issues biologiques, incluant la promotion de la prolifération cellulaire, de la différenciation cellulaire, de l’apoptose, de la migration, de la survie cellulaire en fonction de la kinase et des substrats correspondants (figure 2.8) (Osaki et Gama, 2013).
Par exemple, un signal (p. ex., cytokine) peut activer la MAPKKK, la kinase qui régule le signal d’apoptose (ASK1) (Ichijo et coll., 1997) (figure 2.8). Une série d’événements de phosphorylation s’ensuit, comme la phosphorylation de la MKK3 et de la MKK6 (deux MAPKK). Les deux MAPKK phosphorylées peuvent activer la p38 (une MAPK) dans le cadre d’événements de phosphorylation. La p38 activée cible un certain nombre de substrats, comme le facteur de transcription activant 2 (ATF2), la p53, le STAT1 et l’Elk-1, ce qui entraîne des réponses cellulaires différentes (figure 2.8). La phosphorylation de sérine ciblée par la p38 du suppresseur de tumeur p53 suscite une réponse cellulaire : l’apoptose (Bulavin et coll., 1999).
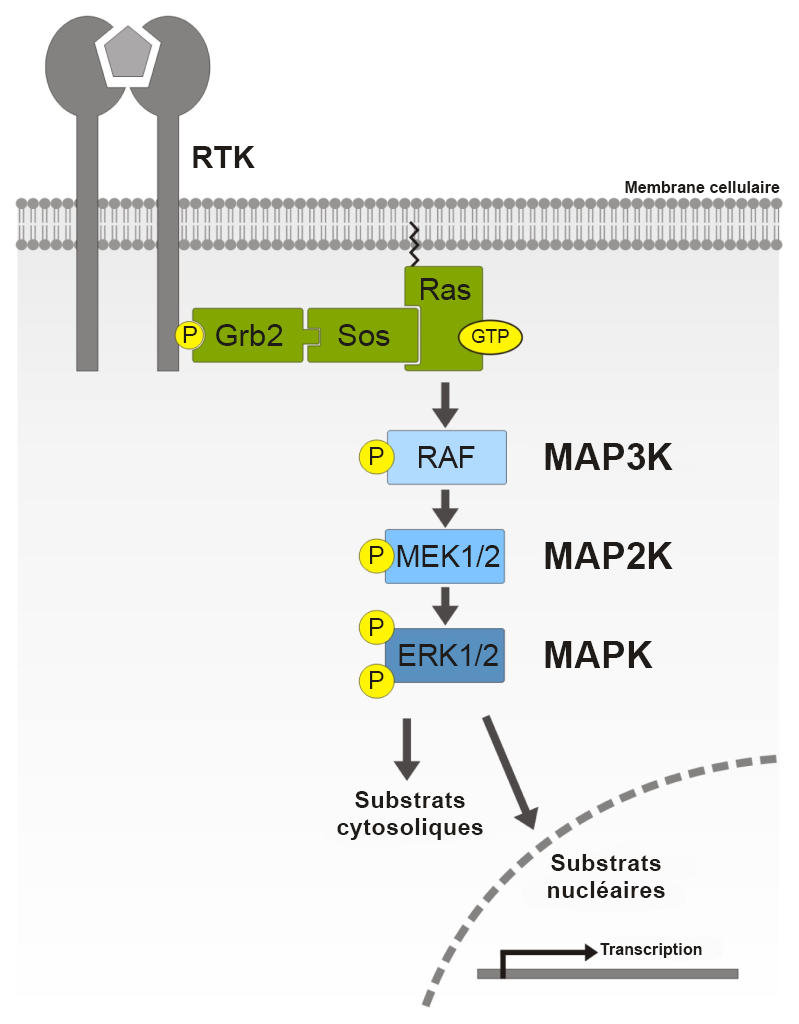
L’IRS à l’état phosphorylé recrute la Grb2 (protéine liée au récepteur du facteur de croissance 2). La Grb2 se lie à l’IRS-1 par le biais du domaine SH2 de la Grb2. Ensuite, la Grb2 recrute la protéine sos. La protéine sos est un GEF qui favorise le passage entre l’état de liaison à la GDP et l’état de liaison à la GTP dans les protéines G. L’une des principales protéines auxquelles s’associe la sos est Ras, qui est converti à l’état de liaison à la GTP.
Rappel : Le Ras-GTP est maintenant à l’état actif et la Ras peut activer la MAPKKK (RAF1) (figure 2.7). Cette activation entraîne une série d’étapes de phosphorylation faisant partie de la cascade MAPK.
Cette interaction est médiée par une « ancre » composée :
a. D’une chaîne polypeptidique
b. D’un groupe lipidique hydrophobe (p. ex. groupement farnésyl)
c. D’une étiquette ubiquitine
d. D’un groupe sucre hydrophile
Faisant partie de la voie MAPK, la Ras est liée à la membrane cellulaire et est située du côté cytosolique à l’aide d’une ancre lipidique appelée groupe farnésyl. L’ancre est illustrée par la structure rouge intégrée à la membrane cellulaire (grise) (figure 2.9) située au niveau de la région C-terminale (Zhou et coll., 2018).
a. sos
b. Ras
c. IR
d. Grb2
La sos est un exemple de facteur d’échange nucléotidiques (GEF).
Dans les voies de signalisation, les protéines adaptatrices permettent de médier les interactions entre les autres protéines. Bien que les protéines adaptatrices contiennent différents domaines protéiques (p. ex., domaines SH2), l’activité catalytique n’est généralement pas attribuée aux protéines adaptatrices. La Grb2 est un exemple de protéine adaptatrice qui participe à plusieurs voies de signalisation, incluant la voie de signalisation de l’insuline. La Grb2 contient un domaine SH2 et deux domaines SH3. Le domaine SH2 sert à lier une molécule de phosphotyrosine activée et les domaines SH3 reconnaissent les séquences riches en proline. En tant que protéine adaptatrice, la Grb2 sert de lien entre la protéine sos (par le biais des domaines SH3) et la protéine RTK (par le biais du domaine SH2).
a. Arrêt de la voie MAPK
b. Activation de l’activité MAPK
c. Aucun effet sur la voie
La sos est une protéine cytosolique. La Grb2 se lie aux domaines riches en proline de la sos par l’intermédiaire des deux domaines SH3 (Buday et coll., 1994). À la suite de ces interactions, la sos se place du côté de la membrane cellulaire près de la Ras (liée au lipide), ce qui favorise l’échange de nucléotides. Toutefois, dans certaines situations, la phosphorylation de la sos perturbe le complexe qui unit la sos à la Grb2, provoquant ainsi la dissociation de ces protéines. Par la suite, cela finira par réguler négativement la voie MAPK (S. Corbalan-Garcia S.-S. Yang et Bar-Sagi, 1996).
Pour mettre en valeur le rôle des protéines d’échafaudage, pensez au rôle des échafaudages lors de la construction de grands immeubles. Le matériel d’échafaudage offre du support pendant le processus d’assemblage. Par analogie, la KSR est une protéine d’échafaudage, offrant aux différentes kinases (p. ex., RAF, MEK, MAPK) une surface sur laquelle elles peuvent s’assembler et se positionner très près les unes des autres (Morrison, 2001).
Les deux catégories de récepteurs sont capables d’effectuer la phosphorylation de la tyrosine et possèdent des domaines intracellulaires, extracellulaires et transmembranaires. Cependant, les RTK abritent directement les domaines kinases dans les régions cytosoliques, tandis que les récepteurs associés à la tyrosine kinase s’associent à une protéine kinase indépendante. L’activité catalytique enzymatique est distincte du domaine C-terminal du récepteur dans les récepteurs associés à la tyrosine kinase.
a. Par covalence
b. De façon non covalente
Les domaines tyrosine kinase ne font pas directement partie de la séquence primaire du récepteur et sont des entités distinctes. Par exemple, les récepteurs de cytokine n’ont pas de domaines tyrosine kinase endogènes. L’activité de la tyrosine kinase est plutôt associée de façon non covalente aux janus kinases (JAK).
Les cytokines sont les molécules de signalisation qui activent les récepteurs de cytokine correspondants. Ce sont des protéines qui favorisent souvent la croissance cellulaire, l’hématopoïèse et les réponses immunitaires. Il y a des divisions floues entre les cytokines et les hormones. Par exemple, une hormone de croissance est souvent considérée comme une cytokine.
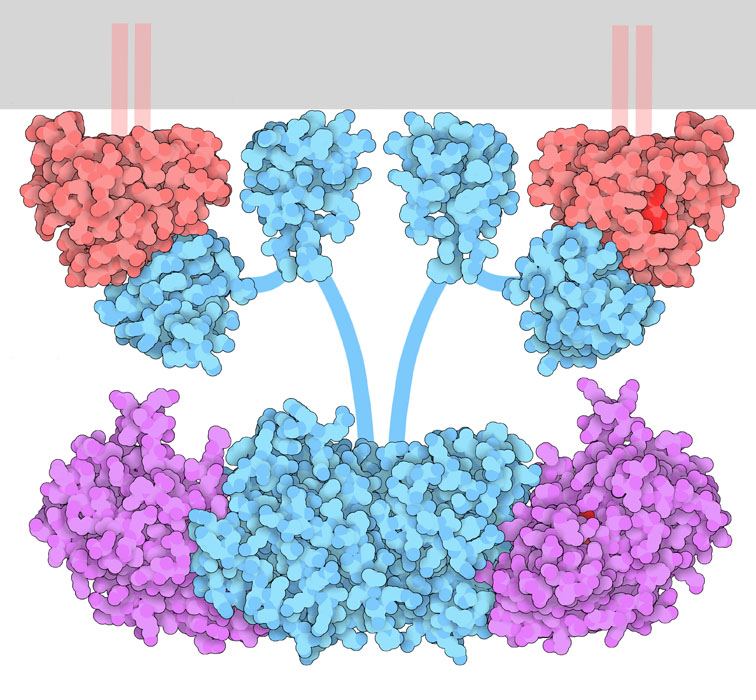
Les récepteurs de cytokine existent généralement sous forme d’une seule unité ou de monomères. La liaison de la cytokine au récepteur entraîne un changement conformationnel qui permet la formation d’homodimères (si les récepteurs sont identiques). Si deux différents types de récepteurs ou plus se dimérisent, il s’agit de ce qu’on appelle l’hétérodimérisation des récepteurs.
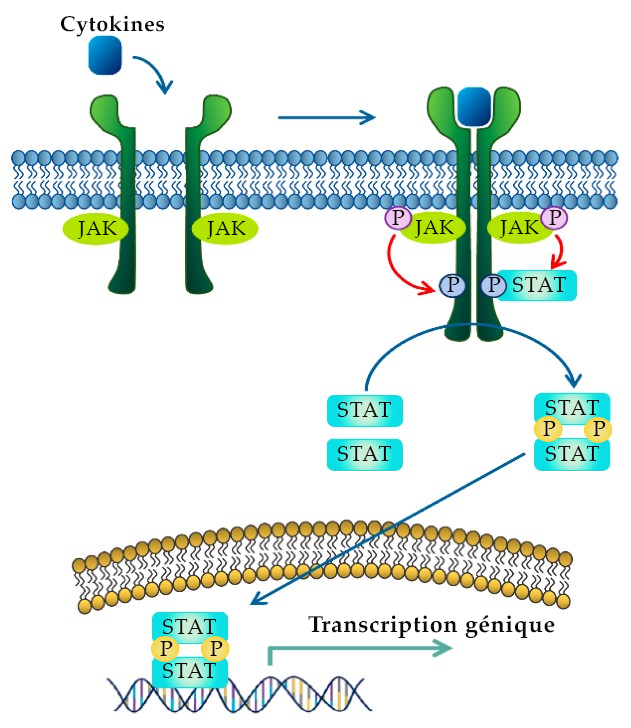
Le mécanisme d’activation des récepteurs de cytokine est semblable à celui des RTK, en ce sens que la liaison du ligand entraîne un changement conformationnel, ce qui amène les monomères à s’approcher les uns des autres. Les tyrosines kinases intracellulaires associées à chaque récepteur seront orientées de façon à phosphoryler la kinase opposée.
Par exemple, la dimérisation du récepteur de cytokine positionne les JAK sur le récepteur de manière à effectuer la trans-autophosphorylation, où ces derniers phosphorylent une tyrosine sur le récepteur opposé. Cette phosphorylation active le JAK, qui phosphoryle ensuite plusieurs sites sur l’extrémité C-terminale de chaque récepteur.
À la suite de la phosphorylation des extrémités C-terminales du récepteur de cytokine, la phosphotyrosine sert de site de liaison à différentes protéines, dont les protéines transducteurs de signaux et activatrices de transcription (STAT). Les protéines STAT sont des facteurs de transcription qui contiennent plusieurs domaines, dont les domaines SH2. Les domaines SH2 remplissent deux fonctions. Au départ, ces domaines reconnaissent les sites de phosphotyrosine sur les extrémités C-terminales cytoplasmiques du récepteur de cytokine et s’y lient. Après l’ancrage des protéines STAT sur le récepteur, le JAK phosphoryler par la suite l’extrémité C-terminale des protéines STAT (figure 2.10). Ces événements de phosphorylation entraînent un changement conformationnel, où les protéines STAT se séparent du récepteur de cytokine. Par conséquent, la phosphotyrosine d’un monomère STAT se lie au domaine SH2 d’une autre protéine STAT, et vice versa, créant un homodimère. L’homodimère STAT pénètre dans le noyau et se lie aux séquences d’ADN de régulation permettant la transcription des gènes ciblés (Erdogan et coll., 2022).
Les domaines SH2 se lient aux motifs contenant de la phosphotyrosine. Des mutations spécifiques dans le domaine SH2 peuvent entraîner une distorsion de la structure secondaire de la protéine. Certaines de ces distorsions peuvent nuire à la structure protéique, ce qui entraîne une perte de fonction. Par exemple, une mutation K591E dans le domaine SH2 du STAT3 entraîne une diminution de l’activité du STAT3 puisque la chaîne latérale de la lysine chargée positivement est remplacée par un acide glutamique chargé négativement (de Araujo, Orccino et coll., 2019). Puisque cette chaîne latérale du résidu coordonne directement la liaison phospho-tyrosine chargée négativement, elle entraîne une répulsion coulombienne et une réduction de l’activité. De la même manière, une mutation N642H du STAT5 entraîne un gain de fonction de la protéine et ce mutant est présent dans plusieurs cancers du sang (de Araujo, Erdogan et coll., 2019; de Araujo et coll., 2020). Cette mutation augmente directement l’affinité du domaine SH2 pour la phosphotyrosine en coordonnant le groupement phosphate, ce qui entraîne finalement des interactions plus étroites avec le récepteur de cytokine, ainsi qu’une prolongation de la durée de vie du dimère du STAT et de l’activité transcriptionnelle.
a. Les protéines STAT se diffusent par le complexe du pore nucléaire.
b. Les protéines STAT nécessitent l’aide des importines.
c. Les STAT ne peuvent pas pénétrer dans le noyau.
Le noyau possède des complexes du pore nucléaire, qui sont intercalés dans la membrane nucléaire, permettant la diffusion de petites molécules (moins de 60 kDa). Les STAT nécessitent l’aide des récepteurs de transport nucléaire, les importines, pour médier l’entrée dans le noyau (figure 2.11) (Reich, 2013).
Contrairement aux RTK, les récepteurs sérine-thréonine kinase (parfois appelés RSTK) font partie de la catégorie des récepteurs couplés à des enzymes qui, en l’occurrence, phosphorylent les résidus de sérine et de thréonine sur les protéines cibles.
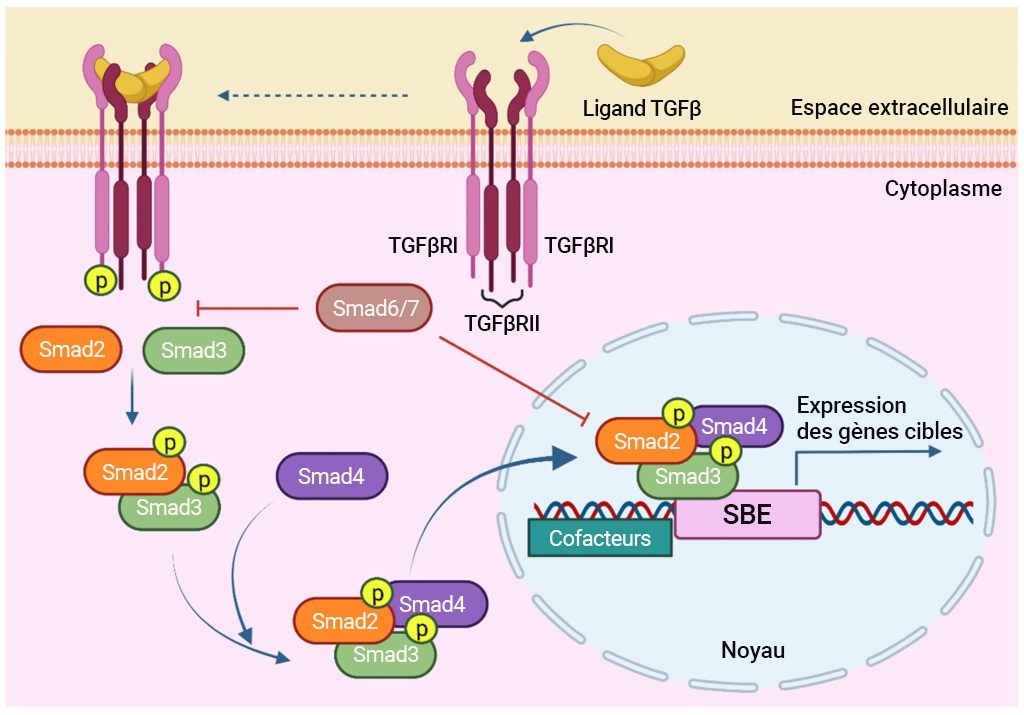
L’exemple prototypique des RSTK est celui des récepteurs du facteur de croissance transformant β (TGFβ) qui peuvent exister sous forme de type I et de type II. Les deux types ont des domaines à membrane unique, ainsi que des domaines intracellulaires et extracellulaires. Les ligands se lient aux domaines extracellulaires des récepteurs homodimères de type II et s’hétérodimérisent avec des récepteurs de type I pour former un complexe hétérotétramère. Le domaine sérine-thréonine kinase intracellulaire exécute une transphosphorylation. Des modifications ou des variations supplémentaires dans la partie C-terminale permettent d’ajouter des couches de régulation.
Bien que les deux récepteurs possèdent des domaines sérine-thréonine kinase, le type I possède un « domaine GS » supplémentaire (Gomperts et coll., 2009). Ce domaine est riche en glycine-sérine, qui peut également être phosphorylée. Les récepteurs de type II possèdent un domaine extracellulaire riche en cystéine.
Le TGFβRII existe en tant qu’homodimère et le ligand (TGFβ) se lie au récepteur (TGFβRII), ce qui entraîne un changement conformationnel. Le TGFβRII se lie au TGFβRI, ce qui permet la phosphorylation de la sérine. Plus précisément, le TGFβII phosphoryle la région riche en sérine du récepteur du domaine GS du TGFβI (Tzavlaki et Moustakas, 2020). Une protéine chaperonne (protéine de liaison FK506 de 12 kilodaltons, la FKBP12) est également associée au TGFβRI. Les changements conformationnels liés à la phosphorylation de la sérine et la libération simultanée de la FKBP12 entraînent l’activation du TGFβRI. À ce stade, il se forme un complexe de récepteurs tétramères actifs constitué de deux récepteurs TGFβ de type II et de deux récepteurs TGFβ de type II (figure 2.12). La liaison au ligand par des molécules telles que le TGFβ, l’activine, les molécules nodales, peut entraîner une association du TGFβ avec les Smad2 (Suppressor of Mothers Against Decapentaplegic) et Smad3, tandis que des ligands tels que la protéine morphogénétique osseuse (BMP) peuvent recruter les Smad1, 5, 8 ou 9. Dans les deux cas, la liaison des Smad est également médiée par une protéine membranaire appelée SARA et la protéine Smad est phosphorylée par le TGFβ. La phosphorylation des Smad entraîne la dissociation de la liaison phospho-Smad et de la protéine SARA. La liaison phospho-Smad forme un complexe avec une autre protéine, la Smad4 (figure 2.12). Ensemble, le complexe phosphorylé Smad-Smad4 pénètre dans le noyau pour réguler l’expression génique.
Les Smad font référence à un groupe de protéines qui sont des facteurs de transcription intervenant dans la régulation de la croissance cellulaire. Les Smad servent de lien entre le signal extracellulaire (TGFβ) et une réponse dans le noyau. Trois membres de la famille Smad interviennent dans la voie TGFβ :
i. R-Smad (comprend Smad1, 2, 3, 5, 8/9)
ii. Co-Smad (Smad médiateurs communs et Smad4 inclus)
iii. I-Smad (Smad inhibiteurs qui suppriment l’action des R-Smad dans le noyau et comprend les Smad6, 7)
a. En présence du ligand TGFβ, le Smad3 est transporté dans le noyau.
b. En présence du ligand TGFβ, le Smad3 reste dans le cytoplasme.
La partie N-terminale de R-Smad contient des domaines, dont un domaine « Mad-homology 1 » (MH1). Une partie du domaine MH1 comprend un NLS (Hill, 2009; Xiao et coll., 2000). Le signal de localisation nucléaire (NLS) est exposé à la phosphorylation. Les protéines ciblées dans le noyau possèdent un NLS au niveau N-terminal, et une version mutante de Smad3, qui possède un NLS non fonctionnel, demeurera dans le cytoplasme.
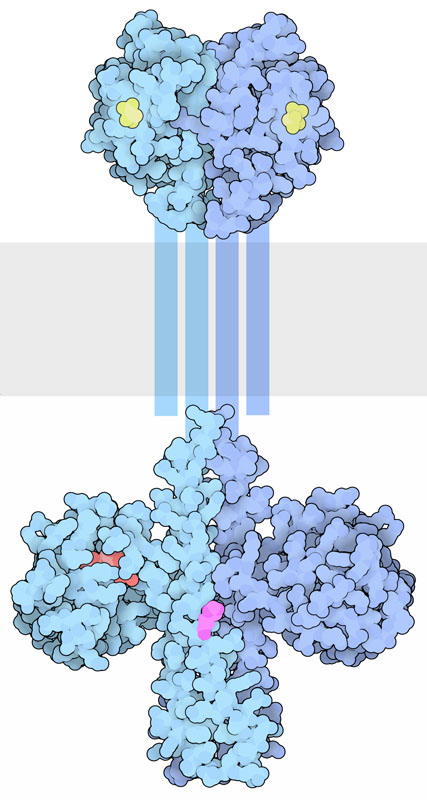
Les protéines transmembranaires de l’histidine kinase se dimérisent au moment de l’activation. La partie extracellulaire contient des domaines de liaison au ligand, tandis que la région cytoplasmique contient un domaine DHp (Dimerization and histidine-phosphotransfer) ainsi que le domaine catalytique (Bhate et coll., 2015; Buschiazzo et Trajtenberg, 2019). Les domaines DHp hébergent une région de la boîte H contenant l’histidine conservée qui est phosphorylée dans le groupe imidazole (figure 2.13). Ce groupement phosphate sur l’histidine est transféré à un aspartate (Asp) sur une autre protéine, ce qui active le régulateur de la réponse protéique.
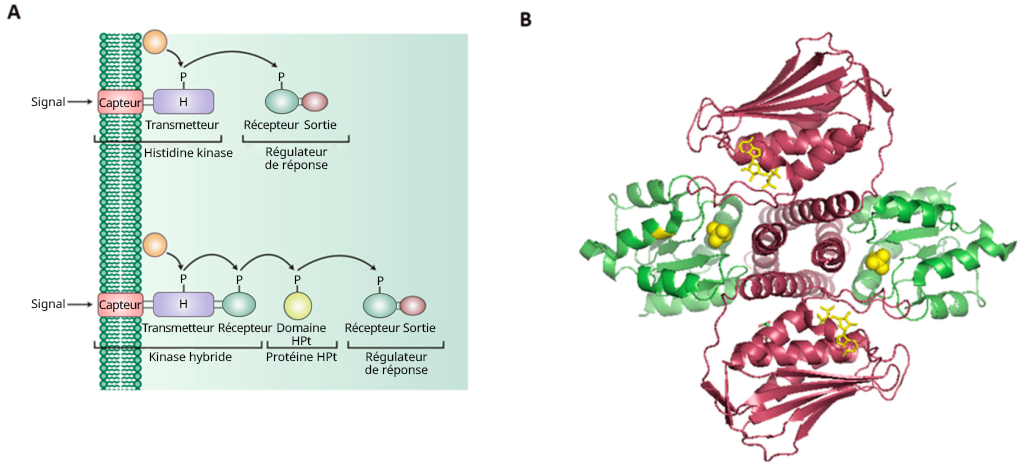
Les récepteurs associés à l’histidine kinase font partie d’un système de signalisation cellulaire à « deux composants ».
Le système à deux composantes comprend deux éléments protéiques :
I. Capteur (contenant un domaine d’entrée [pour la liaison au ligand] et un domaine émetteur [contenant l’histidine conservée et le domaine de liaison de l’ATP et de la kinase]
II. Régulateur de réponse (contenant un domaine récepteur avec l’aspartate conservé et le domaine effecteur ou de sortie)
Ce système est particulièrement important dans les bactéries, pour répondre aux événements extracellulaires, et médie la chimiotaxie.
Ce système est appelé voie de phosphorelais. L’histidine kinase est le capteur, tandis que la protéine effectrice est un régulateur (figure 2.14). Lors de la liaison au ligand, le récepteur (c.-à-d. le capteur) est alors activé, suivi de la trans-phosphorylation d’un résidu d’histidine conservé. Le régulateur de réponse catalyse ensuite le transfert du phosphate du capteur vers un aspartate précis (dans le domaine récepteur du régulateur de réponse) (Papon et Stock, 2019). La phosphorylation de l’aspartate active la protéine effectrice, ce qui entraîne la localisation de l’ADN et la régulation à la hausse de la transcription. En plus
À l’instar d’autres récepteurs tels que les RTK, les récepteurs guanylyl cyclase possèdent des régions extracellulaires et intracellulaires entourant un domaine transmembranaire. Le domaine transmembranaire est constitué d’un segment alpha hélicoïdal, tandis que la région extracellulaire N-terminale contient un domaine de liaison au ligand, et la région C-terminale contient un domaine guanylyl cyclase catalytique. L’activité enzymatique de la guanylyl cyclase convertit la GTP en une version cyclisée, la guanosine monophosphate 3’-5’ cyclique (cGMP) (figure 2.15).
a. La production de la cGMP diminuerait.
b. La production de la cGMP augmenterait.
c. Aucun effet sur la production de la cGMP.
L’enzyme phosphodiestérase catalyse la conversion de la cGMP en 5’-GMP. Le nucléotide cGMP est un exemple de second messager (figure 2.15). Les niveaux de cGMP diminuent en présence de phosphodiestérase.
Il convient de noter que la cGMP forme un complexe avec la protéine kinase G (PKG) dépendante de la cGMP. La PKG est activée et phosphoryle des protéines cibles, potentiellement sur les résidus de sérine-thréonine.
Comme leurs homologues (RTK), les protéines tyrosine phosphatase de type récepteur sont intégrées dans la membrane cellulaire avec d’autres domaines dans l’environnement extracellulaire et dans le cytosol. La région extracellulaire est variable et contient des séquences semblables à celles des motifs de type immunoglobuline, des domaines d’anhydrase carbonique ou des domaines semblables à la fibronectine de type III (Xu et Fisher, 2012). Le domaine de la protéine tyrosine phosphatase (PTP) se trouve dans la région C-terminale et se situe dans le cytosol. Il pourrait y avoir une ou deux copies du domaine PTP, qui comprennent un résidu de cystéine important, essentiel à la déphosphorylation, et le domaine PTP proximal à la membrane est catalytiquement actif, le deuxième domaine affichant des rôles de régulation.
De façon générale, les tyrosines phosphatases de type récepteur effectuent la déphosphorylation des résidus de tyrosine sur les protéines cibles (p. ex., récepteurs avec sites de phosphotyrosine) qui s’oppose aux effets des kinases. Par exemple, la voie MAPK démontre l’équilibrage entre les kinases et les phosphatases. Le récepteur de la protéine tyrosine phosphatase de type R (PTPRR) se lie à la protéine Erk (un MAPK). La protéine Erk est phosphorylée sur la tyrosine et la thréonine, ce qui entraîne l’activation de la kinase. Le PTPRR peut contrer l’activité de kinase de la protéine Erk en supprimant les groupements phosphate (Xu et Fisher, 2012) qui modulent l’activité de la protéine kinase Erk.
a. L’activité de la tyrosine phosphatase de type récepteur serait inhibée.
b. L’activité de la tyrosine phosphatase de type récepteur serait activée.
Dans des conditions d’oxydation, si le site actif de la cystéine est converti en acide sulfonique, il ne sera plus en mesure d’agir comme nucléophile puissant et d’éliminer le groupement phosphate (Xu et Fisher, 2012). On prévoit que l’activité des tyrosines phosphatases de type récepteur serait inhibée.
Alberts, Bruce. (2002). Molecular biology of the cell. Dans Molecular biology of the cell (4e éd.). Garland Science. https://www.ncbi.nlm.nih.gov/books/NBK26822/
Bajusz, D., Pándy-Szekeres, G., Takács, Á., de Araujo, E. D., Erdogan, F., Neubauer, H. A., Meneksedag-Erol,raujo, E. D., et Keserű, G. M. (2023). SH2db, an information system for the SH2 domain. Nucleic Acids Research, 51(W1), W542-W552. https://doi.org/10.1093/nar/gkad420
Bhate, M. P., Molnar, K. S., Goulian, M., et DeGrado, W. F. (2015). Signal Transduction in Histidine Kinases: Insights from New Structures. Structure, 23(6), 981-994. https://doi.org/10.1016/j.str.2015.04.002
Bhagirath, A. Y., Li, Y., Patidar, R., Yerex, K., Ma, X., Kumar, A., et Duan, K. (2019). Two Component Regulatory Systems and Antibiotic Resistance in Gram-Negative Pathogens. International Journal of Molecular Sciences, 20(7). https://doi.org/10.3390/ijms20071781
Buday, L., Egan, S. E., Rodriguez Viciana, P., Cantrell, D. A., et Downward, J. (1994). A complex of Grb2 adaptor protein, Sos exchange factor, and a 36-kDa membrane-bound tyrosine phosphoprotein is implicated in ras activation in T cells. Journal of Biological Chemistry, 269(12), 9019-9023. https://doi.org/10.1016/S0021-9258(17)37070-9
Bulavin, D. V, Saito, S., Hollander, M. C., Sakaguchi, K., Anderson, C. W., Appella, E., et Fornace, A. J. (1999). Phosphorylation of human p53 by p38 kinase coordinates N‐terminal phosphorylation and apoptosis in response to UV radiation. The EMBO Journal, 18(23), 6845-6854. https://doi.org/10.1093/emboj/18.23.6845
Buschiazzo, A., et Trajtenberg, F. (2019). Two-Component Sensing and Regulation: How Do Histidine Kinases Talk with Response Regulators at the Molecular Level? Annual Review of Microbiology, 73(1), 507-528. https://doi.org/10.1146/annurev-micro-091018-054627
de Araujo, E. D., Erdogan, F., Neubauer, H. A., Meneksedag-Erol, D., Manaswiyoungkul, P., Eram, M. S., Seo, H. S., Qadree, A. K., Israelian, J., Orlova, A., Suske, T., Pham, H. T. T., Boersma, A., Tangermann, S., Kenner, L., Rülicke, T., Dong, A., Ravichandran, M., Brown, P. J., … Gunning, P. T. (2019). Structural and functional consequences of the STAT5BN642H driver mutation. Nature Communications, 10(1). https://doi.org/10.1038/s41467-019-10422-7
de Araujo, E. D., Keserű, G. M., Gunning, P. T., et Moriggl, R. (2020).Targeting STAT3 and STAT5 in Cancer. Cancers, 12(8). https://doi.org/10.3390/cancers12082002
de Araujo, E. D., Orlova, A., Neubauer, H. A., Bajusz, D., Seo, H.-S., Dhe-Paganon, S., Keserű, G. M., Moriggl, R., et Gunning, P. T. (2019). Structural Implications of STAT3 and STAT5 SH2 Domain Mutations. Cancers, 11(11). https://doi.org/10.3390/cancers11111757
Erdogan, F., Radu, T. B., Orlova, A., Qadree, A. K., de Araujo, E. D., Israelian, J., Valent, P., Mustjoki, S. M., Herling, M., Moriggl, R., et Gunning, P. T. (2022). JAK-STAT core cancer pathway: An integrative cancer interactome analysis. Journal of Cellular and Molecular Medicine, 26(7), 2049-2062. https://doi.org/10.1111/jcmm.17228
Gomperts, B. D., Kramer, I. M., et Tatham, P. E. R. (2009). Signal transduction . In Signal transduction (2e éd.). Elsevier/Academic Press
Gungor, M. Z., Uysal, M., et Senturk, S. (2022). The Bright and the Dark Side of TGF-β Signaling in Hepatocellular Carcinoma: Mechanisms, Dysregulation, and Therapeutic Implications. Cancers, 14(4). https://doi.org/10.3390/cancers14040940
Hill, C. S. (2009). Nucleocytoplasmic shuttling of Smad proteins. Cell Research, 19(1), 36-46. https://doi.org/10.1038/cr.2008.325
Ichijo, H., Nishida, E., Irie, K., ten Dijke, P., Saitoh, M., Moriguchi, T., Takagi, M., Matsumoto, K., Miyazono, K., et Gotoh, Y. (1997). Induction of Apoptosis by ASK1, a Mammalian MAPKKK That Activates SAPK/JNK and p38 Signaling Pathways. Science, 275(5296), 90-94. https://doi.org/10.1126/science.275.5296.90
Lemmon, M. A., et Schlessinger, J. (2010). Cell Signaling by Receptor Tyrosine Kinases. Cell, 141(7), 1117-1134. https://doi.org/10.1016/j.cell.2010.06.011
Meister, M., Tomasovic, A., Banning, A., et Tikkanen, R. (2013). Mitogen-Activated Protein (MAP) Kinase Scaffolding Proteins: A Recount. International Journal of Molecular Sciences, 14(3), 4854-4884. https://doi.org/10.3390/ijms14034854
Moon, S. Y., Kim, K. D., Yoo, J., Lee, J.-H., et Hwangbo, C. (2021). Phytochemicals Targeting JAK–STAT Pathways in Inflammatory Bowel Disease: Insights from Animal Models. Molecules, 26(9). https://doi.org/10.3390/molecules26092824
Morrison, D. K. (2001). KSR: a MAPK scaffold of the Ras pathway? Journal of Cell Science, 114(9), 1609-1612. https://doi.org/10.1242/jcs.114.9.1609
Morrison, D. K. (2012). MAP kinase pathways. Cold Spring Harbor Perspectives in Biology, 4(11), a011254-a011254. https://doi.org/10.1101%2Fcshperspect.a011254.
Osaki, L. H., et Gama, P. (2013). MAPKs and Signal Transduction in the Control of Gastrointestinal Epithelial Cell Proliferation and Differentiation. International Journal of Molecular Sciences, 14(5), 10143-10161. https://doi.org/10.3390/ijms140510143
Papon, N., et Stock, A. M. (2019). Two-component systems. Current Biology, 29(15), R724-R725. https://doi.org/10.1016/j.cub.2019.06.010
Pike, L. J., Eakes, A. T., et Krebs, E. G. (1986). Characterization of affinity-purified insulin receptor/kinase. Effects of dithiothreitol on receptor/kinase function. Journal of Biological Chemistry, 261(8), 3782-3789. https://doi.org/10.1016/S0021-9258(17)35716-2
Reich, N. C. (2013). STATs get their move on. JAK-STAT, 2(4), e27080. https://doi.org/10.4161/jkst.27080
Robinson, D. R., Wu, Y.-M., et Lin, S.-F. (2000). The protein tyrosine kinase family of the human genome: Tyrosine Kinases. Oncogene, 19(49), 5548-5557. https://www.nature.com/articles/1203957
S. Corbalan-Garcia S.-S. Yang, K. R. D., et Bar-Sagi, D. (1996).Identification of the Mitogen-Activated Protein Kinase Phosphorylation Sites on Human Sosl That Regulate Interaction with Grb2. Molecular and Cellular Biology, 16(10), 5674-5682. https://doi.org/10.1128/MCB.16.10.5674
Sayem, A. S. M., Arya, A., Karimian, H., Krishnasamy, N., Ashok Hasamnis, A., et Hossain, C. F. (2018). Action of Phytochemicals on Insulin Signaling Pathways Accelerating Glucose Transporter (GLUT4) Protein Translocation. Molecules, 23(2). https://doi.org/10.3390/molecules23020258
Ségaliny, A. I., Tellez-Gabriel, M., Heymann, M.-F., et Heymann, D. (2015). Receptor tyrosine kinases: Characterisation, mechanism of action and therapeutic interests for bone cancers. Journal of Bone Oncology, 4(1), 1-12. https://doi.org/10.1016/j.jbo.2015.01.001
Tato, I., Bartrons, R., Ventura, F., et Rosa, J. L. (2011). Amino Acids Activate Mammalian Target of Rapamycin Complex 2 (mTORC2) via PI3K/Akt Signaling. The Journal of Biological Chemistry, 286(8), 6128-6142. https://doi.org/10.1074/jbc.m110.166991
Waksman, G., Shoelson, S. E., Pant, N., Cowburn, D., et Kuriyan, J. (1993). Binding of a high affinity phosphotyrosyl peptide to the Src SH2 domain: Crystal structures of the complexed and peptide-free forms. Cell, 72(5), 779-790. https://doi.org/10.1016/0092-8674(93)90405-F
Xiao, Z., Liu, X., Henis, Y. I., et Lodish, H. F. (2000). A Distinct Nuclear Localization Signal in the N Terminus of Smad 3 Determines Its Ligand-Induced Nuclear Translocation. Proceedings of the National Academy of Sciences – PNAS, 97(14), 7853-7858. https://doi.org/10.1073/pnas.97.14.7853
Xu, Y., et Fisher, G. J. (2012). Receptor type protein tyrosine phosphatases (RPTPs) – roles in signal transduction and human disease. Journal of Cell Communication and Signaling, 6(3), 125-138. https://doi.org/10.1007/s12079-012-0171-5
Yoon, M.-S. (2017). The Role of Mammalian Target of Rapamycin (mTOR) in Insulin Signaling. Nutrients, 9(11), 1176-. https://doi.org/10.3390/nu9111176
Zheng, C. F., et Guan, K. L. (1993). Properties of MEKs, the kinases that phosphorylate and activate the extracellular signal-regulated kinases. The Journal of Biological Chemistry, 268(32), 23933-23939. https://www.jbc.org/article/S0021-9258(20)80474-8/pdf
Zhou, Y., Prakash, P., Gorfe, A. A., et Hancock, J. F. (2018). Ras and the Plasma Membrane: A Complicated Relationship. Cold Spring Harbor Perspectives in Medicine, 8(10). https://doi.org/10.1101/cshperspect.a031831
3
Les RCPG sont un groupe de récepteurs qui jouent un rôle physiologique important dans les processus biologiques, répondant à des signaux comme les composés aromatiques, les neurotransmetteurs, les hormones et même la lumière. Ces stimuli sont relayés en réponse à l’intérieur de la cellule. Contrairement aux RTK, les RCPG activent les protéines G (protéines de liaison aux nucléotides de guanine), qui forment également la base de leur nom. Ce module portera sur les aspects structurels et fonctionnels des RCPG.
À la fin de ce chapitre, vous pourrez :
Les RCPG sont des protéines intégrales (semblables aux récepteurs couplés à des enzymes ou aux RTK) qui sont intégrées à la membrane cellulaire. La structure classique des RCPG se compose de sept hélices α transmembranaires (numérotées de H1 à H7) (figure 3.1). Les parties hélicoïdes sont reliées par des boucles extracellulaires ou intracellulaires. Les ligands peuvent coopérer avec un RCPG en se liant à la partie extracellulaire (située près de l’extrémité N-terminale). L’extrémité C-terminale (partie carboxyle), qui est située dans le cytosol, contient également une hélice α (H8) supplémentaire. Il importe de noter que les RCPG sont également associés à un domaine de liaison des protéines G dans la région cytosolique.
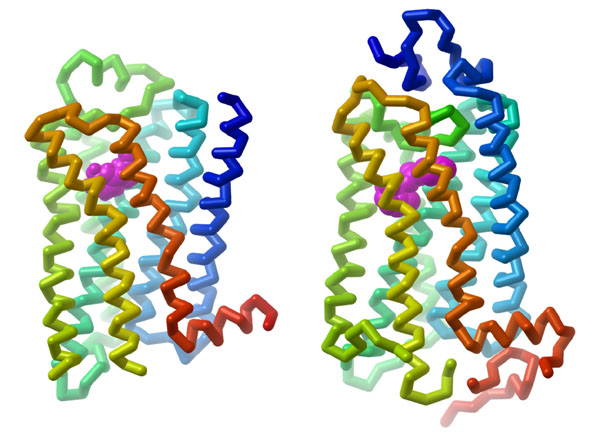
a. Des traitements à l’aide de lipides et de détergents (p. ex., dodécylsulfate de sodium [DSS]).
b. Une incubation avec une solution à faible concentration en sel (p. ex., chlorure de sodium).
c. Une augmentation du pH du tampon alcalin.
Étant donné que les RCPG contiennent plusieurs hélices transmembranaires, ils ont besoin d’un environnement hydrophobe pour préserver leur intégrité structurelle. En raison de la nature des protéines (étroitement associées à la membrane), le traitement à l’aide de détergents ou de bicelles lipidiques peut permettre d’isoler les RCPG (Weis et Kobilka, 2018). Les détergents contenant des régions hydrophobiques peuvent interagir avec la membrane et les hélices transmembranaires. Cette interaction permet d’éliminer le RCPG de la membrane tout en en maintenant la structure globale en la capturant dans une particule contenant des lipides.
a. Radiocristallographie
b. Microscopie par fluorescence
c. Analyse calorimétrique différentielle
La radiocristallographie est une technique biophysique courante qui révèle la structure protéique avec une résolution de niveau atomique. Cette technique utilise un faisceau concentré de rayons X dirigé vers un « cristal » de protéine. Les rayons X sont diffractés lorsqu’ils traversent le cristal et produisent un diagramme de diffraction précis (ou une ombre). Ce diagramme de diffraction peut être déconvolué mathématiquement dans le but de déterminer la position des différents atomes dans le cristal, qui fournit la structure tridimensionnelle de la protéine.
Une protéine se « cristallise » lorsque l’eau est retirée de l’échantillon de protéine d’une manière bien précise. Expérimentalement, ce procédé peut s’avérer extrêmement difficile, mais il s’agit d’une technique puissante qui fournit des images haute résolution des atomes de la protéine. Chaque protéine produit un cristal unique qui dépend de sa structure tridimensionnelle.
Il existe un nombre diversifié de ligands qui peuvent activer les RCPG, notamment des peptides, des nucléotides et des photons bien précis. Toutefois, les RCPG possèdent également une activité endogène, bien qu’elle se produise à un niveau inférieur à celui d’un ligand. Les ligands qui favorisent l’activité des RCPG au-dessus de l’état basal sont appelés agonistes (Weis et Kobilka, 2018). Certains ligands peuvent se lier à un RCPG et bloquer la réponse; c’est alors qu’on les appelle antagonistes.
Le protéome humain contient plus de 900 RCPG et chacun d’eux a un ligand différent (Gaitonde et González-Maeso, 2017). De plus, les RCPG constituent des cibles pour environ 30 % de tous les médicaments offerts sur le marché (Hauser et coll., 2017). Entre autres exemples précis de ligands endogènes du RCPG, on compte l’adrénaline, la noradrénaline, l’histamine, le glucagon, la calcitonine, l’oxytocine et la neurokinine.
Les ligands tels que les peptides se lient à proximité de l’extrémité amino-terminale ou des boucles extracellulaires. Inversement, les petits ligands se lient dans la région transmembranaire des heptahélices (Calebiro et coll., 2021).
Les protéines G sont des protéines de liaison aux nucléotides de guanine qui agissent comme protéines de signalisation intermédiaires entre les RCPG et les seconds messagers. Ces protéines peuvent se lier à la fois à la GTP et au GDP, ce qui détermine si elles sont actives (liées à la GTP) ou inactives (liées au GDP).
Les protéines G peuvent se diviser en deux catégories : i) petites protéines G monomériques et ii) grosses protéines G hétérotrimériques. Les grosses protéines G hétérotrimériques se composent de trois sous-unités protéiques différentes : α (Gα), β (Gβ) et γ (Gγ). Les sous-unités α et γ sont reliées à la membrane par des ancres lipidiques (qui sont de longues queues hydrophobes qui interagissent avec la membrane). Les trois sous-unités existent dans un complexe trimère, y compris une molécule GDP liée à la sous-unité Gα. Lors de l’activation du RCPG (liaison du ligand), le GDP est remplacé par une GTP. Cet échange entraîne la dissociation de la sous-unité Gα des sous-unités Gβ et Gγ. Les sous-unités α et βγ peuvent s’associer à des protéines supplémentaires pour faciliter la transduction de signal.
Les RCPG peuvent également s’associer aux petites protéines G. Ces protéines ressemblent davantage aux sous-unités Gα des protéines G hétérotrimériques et ont également la capacité de se lier à la GTP et au GDP. Les petites protéines G sont généralement reconnues comme des GTPases RAS.
a. La sous-unité Gα pourrait se lier au GMP·PNP, mais elle ne produirait pas d’hydrolyse. Par conséquent, la sous-unité Gα demeurerait à l’état actif.
b. La sous-unité Gα serait en mesure de se lier au GMP·PNP et finalement de se convertir en GMP. Par conséquent, la sous-unité Gα passerait à l’état inactif.
La sous-unité α de la protéine G hétérotrimérique possède un site de liaison du nucléotide guanine. Par conséquent, la sous-unité α peut se lier au GMP·PNP surtout si la concentration est plus élevée que celle de la GTP (Maegley et coll., 1996) (figure 3.2).
Normalement, lorsque le complexe αβγ est lié au GDP, il en résulte un état inactif. Ensemble, les sous-unités βγ stabilisent la sous-unité α à l’état inactif. Lorsque le complexe αβγ est lié à la GTP, il en résulte un état actif, menant finalement à la dissociation de la sous-unité Gα.
a. Gα
b. Gβ
c. Gγ
La sous-unité Gα fonctionne comme une séquence activatrice possédant un domaine GTPase.
a. RCPG
b. Gα
c. Gβ
d. Gγ
Un GEF représente un facteur d’échange des nucléotides guanyliques qui permet le remplacement du GDP par la GTP pour favoriser l’activation du RCPG. Pendant que le RCPG signale la voie, un ligand se lie au récepteur à la surface extracellulaire. Cette action déclenche un changement conformationnel qui se propage à travers les hélices transmembranaires et entraîne l’activité du GEF du côté intracellulaire du RCPG. Par conséquent, le RCPG activé permet de remplacer le GDP par la GTP à la sous-unité Gα, ce qui entraîne l’activation de cette dernière.
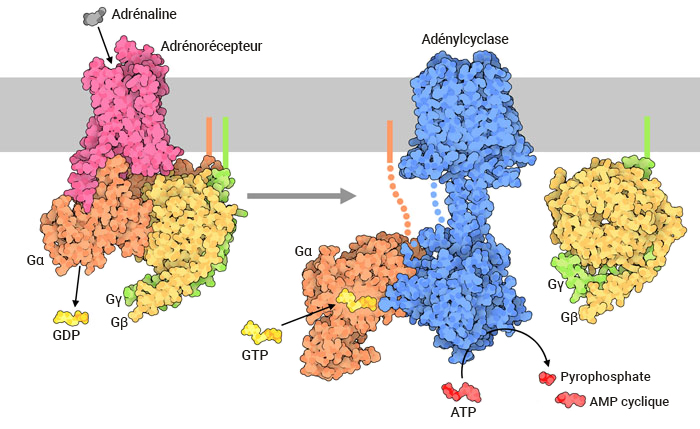
En période de stress, le corps peut réagir en libérant des hormones telles que l’adrénaline (aussi appelée épinéphrine), ce qui permet de venir à bout d’une réponse appropriée aux signaux externes comme la réaction d’attaque-fuite.
Le signal hormonal (adrénaline) se lie à l’adrénorécepteur, ce qui entraîne un changement conformationnel (figure 3.3). Le récepteur s’associe et se lie à la protéine G située dans l’espace cytoplasmique. La sous-unité Gα (illustrée en orange) est associée au GDP, la liaison au RCPG actif déclenche l’activité du GEF et le GDP est remplacé par la GTP. La liaison à la GTP entraîne la dissociation des sous-unités Gα et Gβγ (illustrées en jaune et en vert). La sous-unité Gα dissociée (liée à la GTP) interagit avec un effecteur, comme une kinase, pour stimuler un effet en aval. Dans cette voie, l’effecteur est l’enzyme, l’adénylcyclase (AC), une protéine transmembranaire illustrée en bleu.
L’adénylcyclase catalyse la conversion de l’ATP en AMPc, qui est une puissante molécule de signalisation, appelée second messager (figure 3.3). À mesure qu’augmentent les niveaux du second messager dans le cytoplasme, l’AMPc interagit avec un certain nombre de protéines, dont la protéine kinase A (PKA). Le nucléotide AMPc active la PKA, qui phosphoryle par la suite un certain nombre de protéines cibles, entraînant l’activation de plusieurs protéines participant au métabolisme du glucose et des lipides et fournissant ainsi plus d’énergie au corps.
L’enzyme adénylcyclase, ou adénylate cyclase, est analogue à la guanylyl cyclase. Il catalyse la conversion de l’ATP en version cyclisée (AMP cyclique – AMPc). Une molécule de pyrophosphate est également libérée (figure 3.3).
Le signal extracellulaire est un exemple de premier messager, tandis que les seconds messagers font généralement partie des événements de signalisation en aval. Par exemple, le ligand qui se lie à un RCPG (comme l’adrénaline) est classé comme premier messager. Le premier messager ne traverse généralement pas la membrane cellulaire. Une fois que le premier messager se lie à une cible (comme un RCPG), le signal est transmis à une autre molécule. Dans le cas de l’adrénaline et de l’adrénorécepteur, l’AMPc qui est produite constitue un exemple de second messager. Outre les petites molécules diffusibles, les ions sont un autre exemple courant de seconds messagers. Le processus de conversion d’un signal à un autre est appelé transduction de signal. Ce processus facilite les réponses à différents stimuli. Il importe de noter que le premier messager peut déclencher la production ou la libération de plusieurs molécules de second messager, ce qui amplifie considérablement le signal.
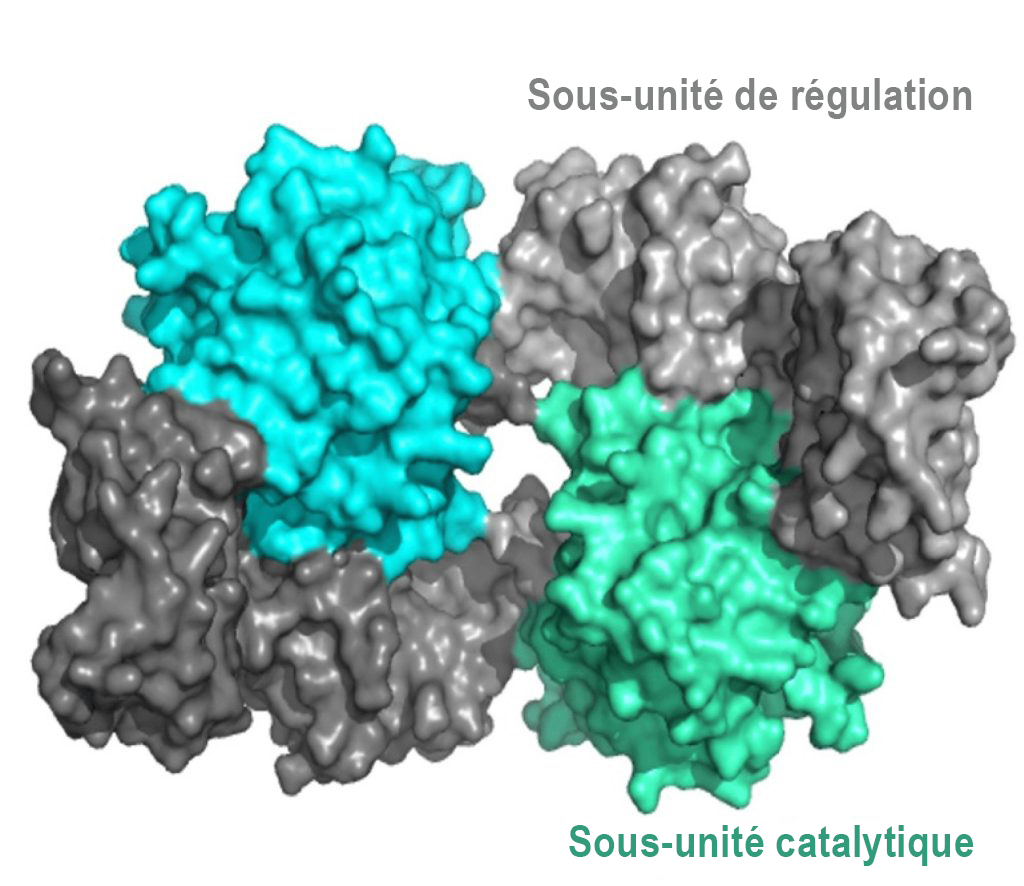
Le mécanisme de régulation servant à l’activation de la PKA fait appel aux molécules AMPc. Structurellement parlant, la PKA est un hétérotétramère composé de deux sous-unités catalytiques et de deux sous-unités de régulation (figure 3.4). Les sous-unités catalytiques hébergent le site actif de la phosphorylation des protéines (et se lient à l’ATP et au substrat), ainsi qu’un domaine de liaison de la sous-unité de régulation. Les sous-unités de régulation possèdent un domaine auto-inhibiteur (qui agit comme un substrat analogique pour se lier à l’activité kinase de la sous-unité catalytique, et inhiber celle-ci), ainsi qu’un domaine de liaison de l’AMPc et un domaine de dimérisation. Les sous-unités de régulation existent sous différents isoformes, et la dimérisation peut être covalente par l’entremise de ponts disulfures ou non covalente.
En l’absence (ou en raison d’une faible concentration) d’AMPc, le dimère de la sous-unité de régulation est associé aux sous-unités catalytiques de la PKA, qui maintiennent les protéines dans une conformation inactive. En présence d’AMPc (ou de concentrations croissantes), l’AMPc se lie aux sous-unités de régulation du domaine de liaison de l’AMPc, entraînant une modulation allostérique et la dissociation des sous-unités catalytiques. Les sous-unités catalytiques de la PKA qui sont libres peuvent maintenant se lier et phosphoryler les cibles de la cellule en aval.
a. Sérine
b. Thréonine
c. Tyrosine
d. a et b
La PKA est une sérine/thréonine kinase. Cependant, la PKA se situe souvent dans la membrane cellulaire en raison de son association à une protéine d’échafaudage, la protéine kinase A d’ancrage (AKAP) (figure 3.5) (Welsh et coll., 2023). Dans certains cas, cela introduit une couche de ciblage spatial pour la kinase et un autre mécanisme de régulation.
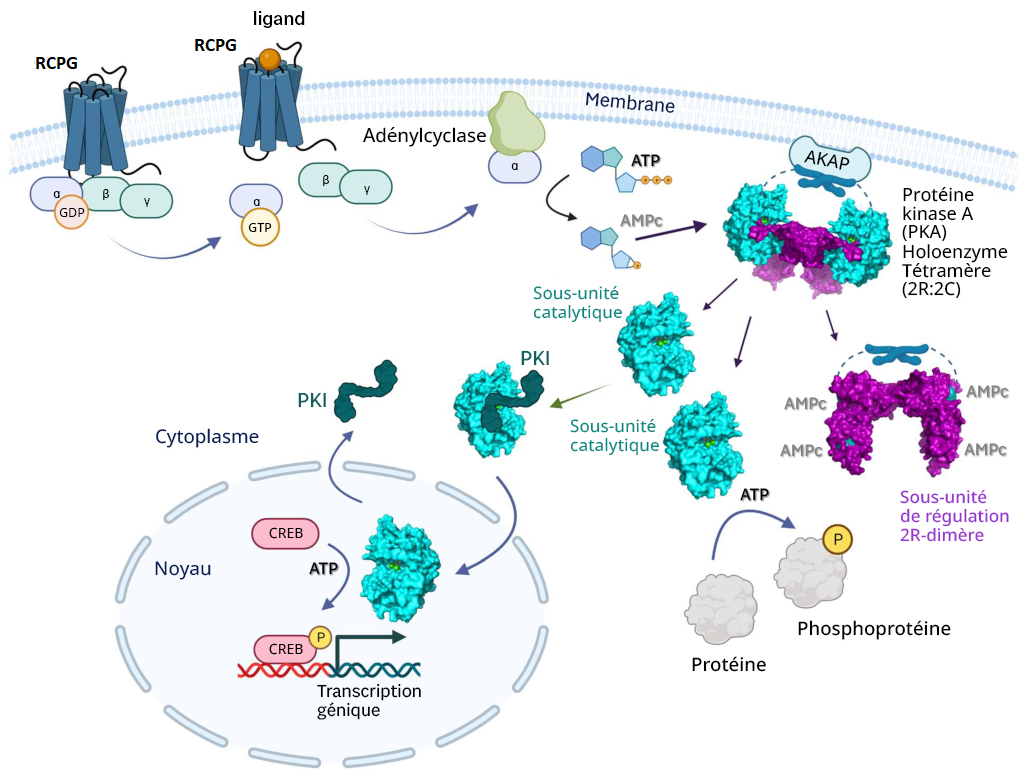
L’adénylcyclase entraîne une augmentation de la production d’AMPc, qui (comme nous l’avons décrit ci-dessus) active la PKA. Il se peut que les sous-unités catalytiques de la PKA ne soient plus ancrées à la membrane (si elles ne sont pas associées aux protéines d’ancrage lipidiques) et qu’elles puissent se diffuser dans d’autres régions de la cellule. Cela comprend la capacité d’entrer dans le noyau et de réguler l’expression génique. À l’intérieur du noyau, la PKA phosphoryle une sérine sur différents facteurs de transcription tels que la protéine se liant au CRE (CREB) (figure 3.5). La CREB peut se lier à une autre protéine CBP (protéine se liant au CREB) et le complexe CREB/CBP se lie au CRE (élément de réponse à l’AMPc), une séquence d’ADN qui flanque les gènes sous son contrôle, y compris les neuropeptides (p. ex., somatostatine, enképhaline), la tyrosine-hydroxylase et les autres protéines impliquées dans les rythmes circadiens (Montminy et coll., 1990).
Il s’agit de protéines en aval de la voie modifiée par le signal. Par exemple, un RCPG n’est généralement pas une protéine effectrice, mais l’adénylcyclase en est une. L’adénylcyclase exécute l’activité en aval et réalise l’« effet » du signal original (événement de liaison au ligand).
Il existe une variété de sous-unités α responsables de différentes réactions physiologiques. Par exemple, les protéines αs (aussi appelées Gαs) stimulent l’enzyme adénylcyclase. Inversement, la sous-unité α de la protéine G inhibitrice (αi ou Gαi) se lie à l’enzyme adénylcyclase, et inhibe celle-ci.
À l’instar de l’adrénorécepteur β (où l’adrénaline active le RCPG), différents ligands (comme l’hormone angiotensine II) se lient et activent différents types de RCPG (comme l’AT1 ou l’AT2). Comme nous l’avons déjà décrit, au moment de la liaison, de l’activation et de l’échange de la GTP par le RCPG, la sous-unité α de la protéine G liée à la GTP se dissocie du complexe βγ. La sous-unité Gα se lie et active l’enzyme PLC ou phospholipase C (la protéine effectrice). La PLC divise la phosphatidylinositol 4,5-bisphosphate (PIP2 ou PI[4,5] P2) en principaux produits, le diacylglycérol (DAG) et l’inositol 1,4,5-trisphosphate (IP3) (Kadamur et Ross, 2013).
Le destin de ces deux produits a des effets importants sur la cellule. L’IP3 se lie aux canaux calciques de libération Ca2+ sensible à l’IP3 sur la membrane du réticulum endoplasmique (RE), ce qui en entraîne l’ouverture Le Ca2+ emmagasiné dans le RE est libéré dans le cytosol et peut se lier à un certain nombre de protéines cibles, dont la protéine kinase C (PKC).
Le DAG est un autre produit découlant de l’action enzymatique de la PLC. Le DAG est intégré dans le feuillet valvulaire interne de la membrane cellulaire. Le DAG active la PKC, qui phosphoryle diverses cibles sur les résidus de la sérine et de la thréonine. La PKC liée au Ca2+ est capable de fonctionner avec le DAG par l’intermédiaire de son domaine de liaison au DAG (figure 3.6).
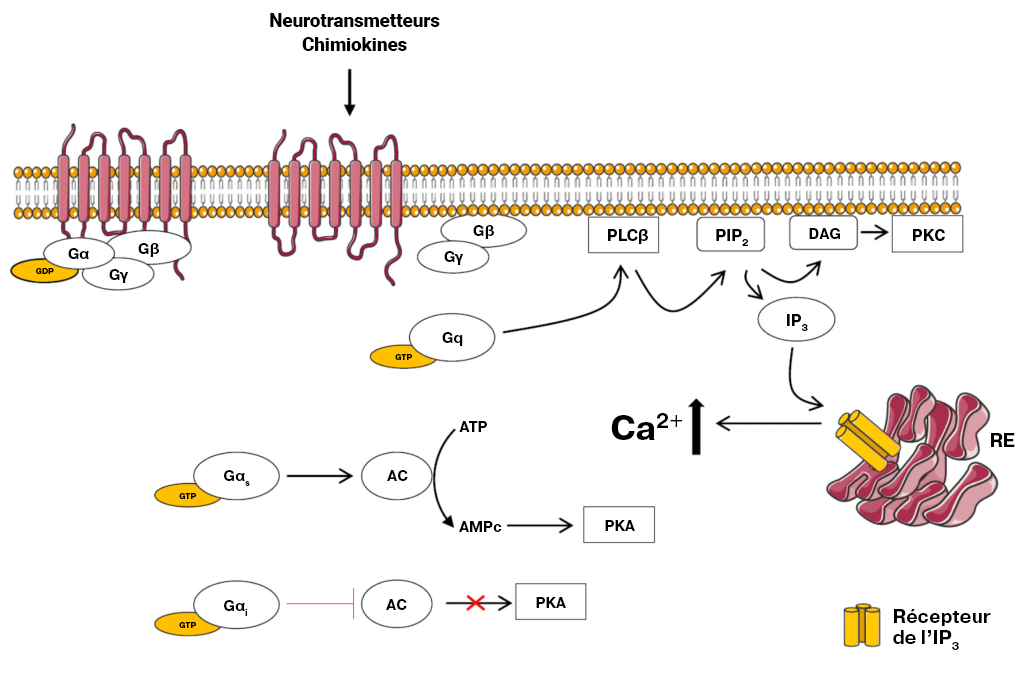
Souvenez-vous que la structure de la membrane cellulaire est une bicouche lipidique composée d’une membrane ou d’un feuillet valvulaire intérieur et extérieur. Le PIP2 se trouve sur le feuillet valvulaire de la membrane intérieure.
a. L’IP3
b. Le DAG
c. a et b
L’IP3 et le DAG sont des exemples de seconds messagers, dont la concentration cellulaire augmente en réaction à l’activation de la voie de signalisation du RCPG.
Cela signifie simplement que les voies de signalisation sont souvent introduites sous forme de voies linéaires qui fonctionnent à la fois de façon séquentielle et isolée. Toutefois, c’est rarement le cas et les voies de signalisation sont souvent interconnectées et entraînent une régulation à la hausse et une régulation à la baisse complexes de nombreuses protéines différentes (p. ex., figure 3.7). L’activité d’une seule cible est souvent régie par l’action consensuelle de nombreuses protéines effectrices différentes. Il existe des activateurs, des inhibiteurs, des chaperons et d’autres protéines qui agissent à titre de régulateurs à différents moments à chacune des étapes biochimiques. Cela procure un degré élevé de régulation afin de contrôler précisément le résultat physiologique provenant de la voie.
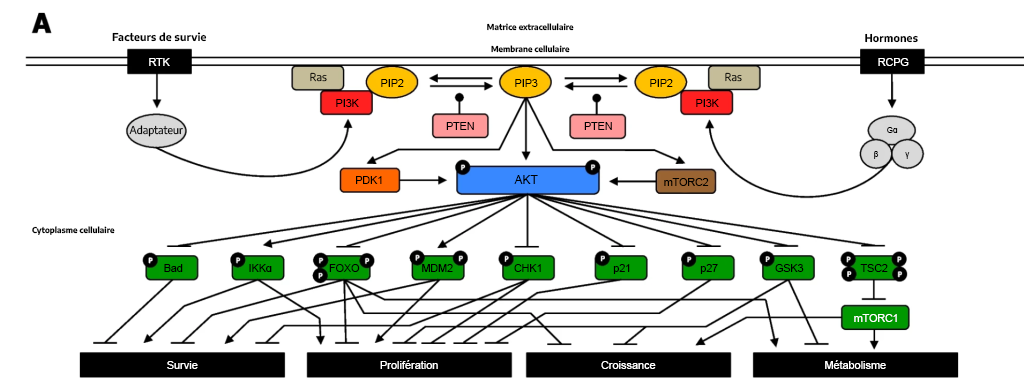
Le symbole P majuscule indique que la protéine est phosphorylée.
a. L’Akt phosphoryle la protéine Bad qui en entraîne l’inhibition.
b. L’Akt inhibe le gène Bad.
c. L’Akt phosphoryle la protéine Bad, ce qui entraîne l’activation de la Bad.
d. L’Akt active le gène Bad.
Une tête de flèche émoussée indique que l’Akt phosphorylé inhibe la protéine Bad, alors qu’une tête de flèche normale indique que la réaction se déroule normalement. L’Akt phosphoryle la protéine Bad, ce qui en entraîne l’inhibition.
a. L’Akt phosphoryle la protéine IKKα, l’inhibant par le fait même.
b. L’Akt inhibe le gène IKKα (gène CHUK).
c. L’Akt phosphoryle la protéine IKKα, l’activant par le fait même.
d. L’Akt active le gène IKKα (gène CHUK).
La flèche indique que l’Akt phosphoryle la protéine IKKα, activant ainsi cette dernière.
Les RTK et les RCPG participent à la régulation de la voie PI3K/Akt. Pour les deux récepteurs, le ligand apparenté peut se lier et induire un changement conformationnel qui aura des répercussions et qui convergera sur les kinases en aval, notamment la PI3K. Dans le cas des RCPG, il activera la PI3K, tandis qu’une cascade de kinases finira par entraîner l’activation de la PI3K dans les RTK.
La phosphorylation de la PI3K entraîne la formation de PIP3. Le PIP3 se lie aux protéines, y compris l’Akt (PKB) et la PDK1. L’Akt des kinases sérine et thréonine phosphoryle un certain nombre de cibles en aval, ce qui en entraîne l’activation ou l’inhibition. Cette action a une incidence sur une pléthore de processus cellulaires. L’Akt phosphoryle un éventail de protéines, comme le promoteur mort associé à Bcl-2 (Bad), les facteurs de transcription FOXO (ou tout simplement FOXO), la protéine Checkpoint kinase 1 (CHK1), le p21, le p27 et le glycogène synthase kinase 3 (GSK3), les inhibant par le fait même (figure 3.7). L’Akt phosphoryle également la protéine, la sclérose tubéreuse de Bourneville du gène TSC2 (ou tout simplement TSC2), ce qui en entraîne l’inhibition, permettant ainsi l’activation du mTORC1. Inversement, la phosphorylation de l’Akt active la IκBα kinase α (IKKα) et la murine double minute-2 (MDM2).
Le phosphatase PTEN élimine les groupes de phosphate du PIP3 pour former le PIP2.
a. Augmentation
b. Diminution
c. Aucune incidence sur les niveaux de phosphorylation
Le bisperoxovanadium est un inhibiteur du PTEN (Schmid et coll., 2004) qui bloquerait l’activité du phosphatase de la protéine. On prévoit que les niveaux de phosphorylation augmenteraient.
Boczek, T., Mackiewicz, J., Sobolczyk, M., Wawrzyniak, J., Lisek, M., Ferenc, B., Guo, F. et Zylinska, L. « The Role of G Protein-Coupled Receptors (GPCRs) and Calcium Signaling in Schizophrenia. Focus on GPCRs Activated by Neurotransmitters and Chemokines », Cells, 10(5). https://doi.org/10.3390/cells10051228
Calebiro, D., Koszegi, Z., Lanoiselée, Y., Miljus, T., et O’Brien, S. (2021). G protein-coupled receptor-G protein interactions: a single-molecule perspective. Physiological Reviews, 101(3), 857-906. https://doi.org/10.1152/physrev.00021.2020
Gaitonde, S. A., et González-Maeso, J. (2017). Contribution of heteromerization to G protein-coupled receptor function. Current Opinion in Pharmacology, 32, 23-31. https://doi.org/10.1016/j.coph.2016.10.006
Glaviano, A., Foo, A. S. C., Lam, H. Y., Yap, K. C. H., Jacot, W., Jones, R. H., Eng, H., Nair, M. G., Makvandi, P., Geoerger, B., Kulke, M. H., Baird, R. D., Prabhu, J. S., Carbone, D., Pecoraro, C., Teh, D. B. L., Sethi, G., Cavalieri, V., Lin, K. H., … Kumar, A. P. (2023). PI3K/AKT/mTOR signaling transduction pathway and targeted therapies in cancer. Molecular Cancer, 22(1), 1-138. https://doi.org/10.1186/s12943-023-01827-6
Hauser, A. S., Attwood, M. M., Rask-Andersen, M., Schiöth, H. B., et Gloriam, D. E. (2017). Trends in GPCR drug discovery: new agents, targets and indications. Nature Reviews. Drug Discovery, 16(12), 829-842. https://doi.org/10.1038/nrd.2017.178
Kadamur, G., et Ross, E. M. (2013). Mammalian phospholipase C. Annual Review of Physiology, 75(1), 127-154. https://doi.org/10.1146/annurev-physiol-030212-183750
Maegley, K. A., Admiraal, S. J., et Herschlag, D. (1996). Ras-Catalyzed Hydrolysis of GTP: A New Perspective from Model Studies. Proceedings of the National Academy of Sciences – PNAS, 93(16), 8160-8166. https://doi.org/10.1073/pnas.93.16.8160
Montminy, M. R., Gonzalez, G. A., et Yamamoto, K. K. (1990). Regulation of camp-inducible genes by creb. Trends in Neurosciences, 13(5), 184-188. https://doi.org/10.1016/0166-2236(90)90045-C
Schmid, A. C., Byrne, R. D., Vilar, R., et Woscholski, R. (2004). Bisperoxovanadium compounds are potent PTEN inhibitors. FEBS Letters, 566(1–3), 35-38. https://doi.org/10.1016/j.febslet.2004.03.102
Seok, S.-H. (2021). Structural Insights into Protein Regulation by Phosphorylation and Substrate Recognition of Protein Kinases/Phosphatases. Life, 11(9). https://doi.org/10.3390/life11090957
Weis, W. I., et Kobilka, B. K. (2018). The Molecular Basis of G Protein–Coupled Receptor Activation. Annual Review of Biochemistry, 87(1), 897-919. https://doi.org/10.1146/annurev-biochem-060614-033910
Welsh, C. L., Conklin, A. E., et Madan, L. K. (2023). Interaction Networks Explain Holoenzyme Allostery in Protein Kinase A. Kinases and Phosphatases, 1(4), 265-287. https://doi.org/10.3390/kinasesphosphatases1040016
4
Les récepteurs nucléaires sont des facteurs de transcription qui jouent un rôle dans l’expression génique influençant des processus comme le métabolisme, la prolifération, l’équilibre électrolytique, la reproduction et l’inflammation (Lavery et McEwan, 2005; Mazaira G. I., 2018). En raison du rôle important qu’ils jouent dans un certain nombre d’activités physiologiques, les récepteurs nucléaires sont également la cible de la thérapeutique. Cette unité vise à décrire le mécanisme d’action des récepteurs nucléaires dans les voies de signalisation. Les aspects structurels et fonctionnels des récepteurs nucléaires seront décrits.
À la fin de ce chapitre, vous pourrez :
a. Ils possèdent des groupes aromatiques
b. Leur composition est principalement hydrophile
c. Leur composition est principalement hydrophobe
d. Options a et b
e. Options a et c
Les ligands qui ciblent les récepteurs nucléaires sont différents de ceux qui ciblent les récepteurs couplés aux enzymes ou les récepteurs couplés aux protéines G (abordés précédemment). Cela s’explique principalement par le fait que ces ligands sont relativement lipophiles et sont capables de traverser passivement la membrane cellulaire et de pénétrer dans le cytosol pour produire leurs effets. Les ligands des récepteurs couplés à des enzymes ou les RCPG sont soit trop gros, soit trop polaires et ne peuvent pas traverser la membrane cellulaire.
Par exemple, la testostérone est un ligand hormonal pour le récepteur nucléaire des androgènes. La testostérone se compose d’un anneau cyclopentanoperhydrophénanthrène (qui est le même noyau stéroïdien que le stérol commun, le cholestérol) (figure 4.1). La structure globale est relativement non polaire, à l’exception de deux atomes d’oxygène terminaux qui se situent à chaque extrémité de la molécule. Cela facilite la liaison de la testostérone à son récepteur nucléaire correspondant, car la pochette de liaison est relativement hydrophobe, à l’exception des chaînes latérales d’Arg752 et de Thr877, qui interagissent respectivement par liaison hydrogène avec les groupes fonctionnels carbonyle et hydroxyle de la testostérone (de Jésus-Tran et coll., 2006).
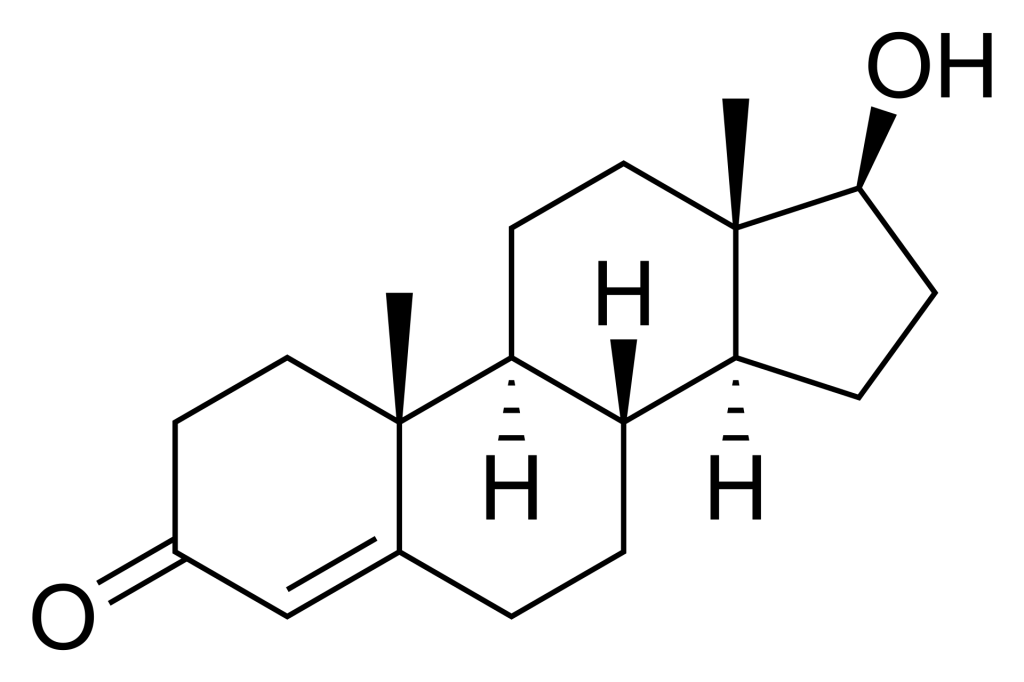
a. Les ligands des récepteurs nucléaires agissent de manière similaire à ceux des récepteurs-enzymes en se liant dans la région extracellulaire et en produisant leurs effets.
b. Les ligands des récepteurs nucléaires traversent la bicouche lipidique de la membrane cellulaire et se lient aux molécules réceptrices pour former un complexe ligand-récepteur, après avoir pénétré dans le noyau.
Comme nous l’avons mentionné précédemment, les ligands qui activent les récepteurs nucléaires sont capables de traverser passivement la membrane cellulaire en raison de leur nature lipophile. Les récepteurs nucléaires se distinguent des autres récepteurs dans la mesure où ce sont des protéines intracellulaires (qui ne sont pas intégrées à la membrane cellulaire). On les appelle également « facteurs de transcription », car ils se lient à l’ADN et facilitent directement la transcription au moment de la liaison du ligand. En fait, les récepteurs nucléaires représentent la plus grande famille de facteurs de transcription eucaryotes (Mazaira G. I., 2018).
Il existe 48 récepteurs nucléaires répertoriés chez les êtres humains (Weikum et coll., 2018). Au cours des 50 dernières années, ces récepteurs ont été classés en différentes catégories en fonction de leur identification historique, de la similarité de leur séquence, de leur réponse au substrat du ligand, de leur dimérisation et de leurs éléments de liaison à l’ADN. Par exemple, les récepteurs nucléaires ont initialement été classés en trois types différents (récepteurs endocriniens, récepteurs orphelins et récepteurs orphelins adoptés), et il existe également des classifications fondées sur la similarité des séquences qui ont conduit à la création de six sous-familles (Laudet, 1997).
Habituellement, la classification la plus simple répertorie deux types de récepteurs nucléaires, appelés récepteurs de type I et de type II. Les récepteurs de type I sont situés dans le cytoplasme et transitent vers le noyau après la liaison de l’hormone. Les récepteurs des androgènes, de la progestérone et des glucocorticoïdes sont des exemples de récepteurs de type I. Les récepteurs de type II se trouvent dans le noyau et sont liés à l’ADN même en l’absence de ligand. Les récepteurs activés par les proliférateurs de peroxysomes (PPAR) et les récepteurs de l’acide rétinoïque (RAR) sont des exemples de récepteurs de type II.
Différentes nomenclatures ont été utilisées pour désigner les récepteurs nucléaires. Dans le cas le plus générique, tous les récepteurs nucléaires contiennent une région de liaison à l’ADN et une région de liaison au ligand. Selon la séquence, les récepteurs nucléaires sont également désignés par les régions A à F (figure 4.2). Ces régions contiennent des domaines spécifiques qui jouent un rôle important sur le plan de l’activité. La région A/B, située à l’extrémité N-terminale, est très peu structurée et contient la fonction d’activation 1 (AF-1). La région C contient un domaine de liaison à l’ADN (DBD) qui joue un rôle important pour lier des séquences spécifiques de l’ADN et amorcer la transcription. La région D sert de boucle de connexion entre le DBD et la région E/F et est parfois appelée région charnière. La région E/F est située à l’extrémité C-terminale et abrite le domaine de liaison au ligand ainsi que le motif de la fonction d’activation 2 (AF-2).
Les DBD permettent aux récepteurs nucléaires de se lier à l’ADN dans des régions spécifiques appelées éléments de réponse. Le DBD contient également deux motifs en doigts de zinc. En plus des différents domaines, les récepteurs nucléaires possèdent différents signaux de localisation nucléaire (NLS) identifiés dans le domaine D (région charnière), qui facilitent la localisation cellulaire dans le noyau (Weikum et coll., 2018).
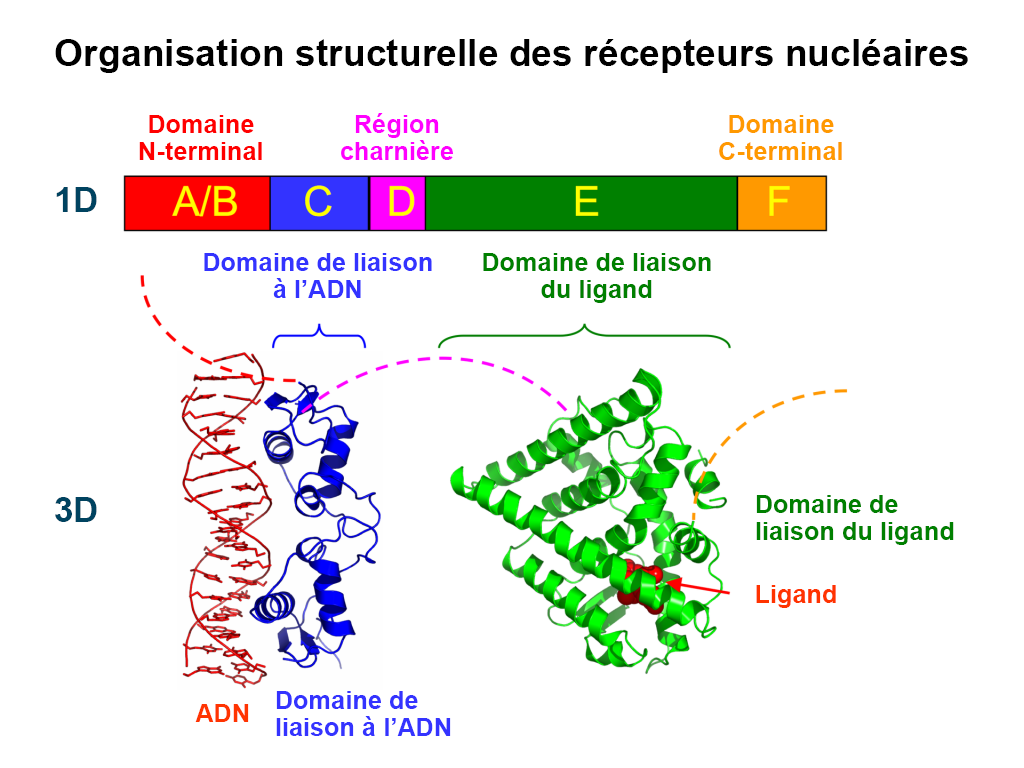
Les ligands se lient à l’extrémité C-terminale des récepteurs nucléaires dans la région E/F ou le domaine de liaison du ligand (LBD). Cette liaison déclenche des changements conformationnels spécifiques dans l’ensemble du domaine de liaison du ligand. Plus précisément, ce domaine se compose de 12 hélices α et d’une pochette non polaire permettant la liaison du ligand (figure 4.2). La dernière hélice (H12) est repositionnée de manière à créer une structure plus condensée et une pochette de surface qui permet des interactions avec la région A/B ainsi qu’avec d’autres protéines corégulatrices. Cela favorise également la dimérisation du récepteur nucléaire, ce qui renforce encore davantage l’activation du récepteur.
Pour les récepteurs de type I, la liaison du ligand se produit (dans le domaine de liaison du ligand) dans le cytosol après la diffusion de l’hormone à travers la membrane cellulaire. L’événement de liaison déclenche des changements conformationnels qui entraînent la dissociation des protéines stabilisatrices comme les protéines de choc thermique, ce qui permet également la dimérisation dans le cytosol. La dimérisation est facilitée par le DBD et le LBD. Les sites AF-1 et AF-2 se lient à d’autres protéines recrutées et qui permettent d’activer le récepteur nucléaire (p. ex., la famille des coactivateurs stéroïdiens Src/p160 – p160). Le complexe situé à l’intérieur du noyau se lie aux éléments de réponse des hormones pour amorcer la transcription (figure 4.3) (Feng et He, 2019).
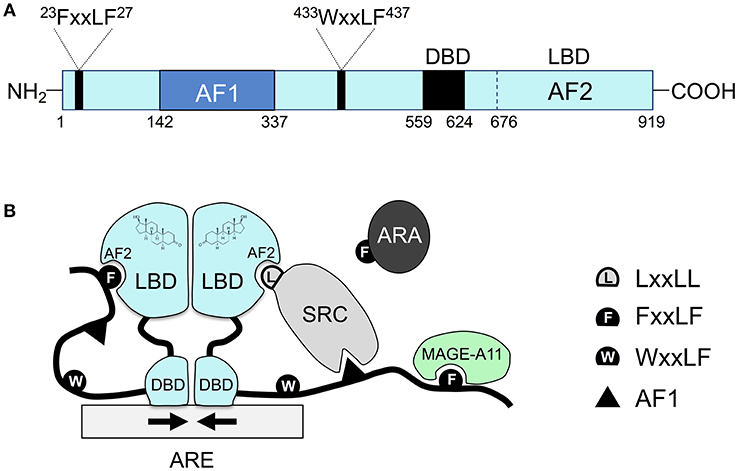
Les récepteurs de type II suivent une voie similaire, sauf que le récepteur nucléaire est toujours lié à l’ADN et forme généralement un complexe avec une protéine corépressive (Sever et Glass, 2013). La liaison du ligand (p. ex., une hormone) libère le corépresseur et active l’assemblage d’un coactivateur et de l’ARN polymérase, ce qui conduit finalement à la transcription des protéines cibles.
Imaginons une situation hypothétique dans laquelle un récepteur nucléaire présente différentes valeurs de Kd (constante de dissociation) pour différents ligands.
a. Kd = 0,5 nmol
b. Kd = 5 nmol
c. Kd = 50 nmol
La réaction d’une protéine (ou d’un récepteur) avec un ligand est exprimée par l’équation : P + L ↔ P-L.
La constante d’association (Ka) peut être exprimée quantitativement comme étant la concentration des produits divisée par la concentration des substrats. Cependant, les unités de cette réaction s’expriment en m-1, ce qui est difficile à comparer intuitivement. C’est pourquoi les constantes de dissociation sont couramment utilisées pour décrire les différentes réactions et fournir une valeur qui peut être facilement interprétée. La valeur de Kd représente la concentration requise pour que la moitié du récepteur ou de la protéine soit saturée. Par conséquent, une valeur de Kd inférieure correspond à un composé plus puissant, puisqu’une concentration plus faible est nécessaire pour interagir avec la cible. Fait important à noter, la constante de dissociation est l’inverse de la constante d’association. Ainsi, dans l’exemple ci-dessus, le substrat ayant une valeur de Kd = 0,5 nmol représente la plus grande affinité. Il importe de préciser que les différents substrats ou ligands peuvent avoir différentes valeurs de Kd pour différents récepteurs. Par exemple, l’estriol (un analogue de l’œstrogène) a une valeur de Kd environ deux fois moins élevée pour le récepteur d’œstrogène alpha que pour le récepteur d’œstrogène bêta (Dahlman-Wright et coll., 2003).
a. Microtubules
b. Microfilaments
c. Filaments intermédiaires
Les complexes ligand-récepteur nucléaire cytosoliques sont acheminés vers les pores nucléaires à l’aide des microtubules, qui constituent la principale voie de circulation (Gomperts et coll., 2009).
Nous avons décrit différents types de récepteurs nucléaires. Dans la majorité de ces scénarios, le ligand et le récepteur ont été définis (p. ex., le cortisol, un stéroïde, se lie au récepteur des glucocorticoïdes). Cependant, il existe des récepteurs nucléaires dont le ligand demeure inconnu. Ces récepteurs nucléaires sont caractérisés comme étant des récepteurs orphelins, mais ils peuvent tout de même avoir une incidence sur les processus de transcription, en les activant ou en les réprimant (Gomperts et coll., 2009). Si le ligand est découvert ultérieurement, ces récepteurs sont mis à jour et deviennent des « récepteurs orphelins adoptés » (Li et coll., 2003). Par exemple, les récepteurs PPAR (alpha, gamma et delta) sont tous considérés comme étant des récepteurs orphelins adoptés puisqu’ils ont été initialement désignés comme des protéines critiques dans le métabolisme cellulaire. On a constaté qu’ils répondent à un certain nombre de substrats (p. ex., PPARα est activé par les acides gras libres) et qu’ils ont des effets pharmacologiques importants pour un certain nombre de maladies comme l’hyperlipidémie et le diabète.
Il a été démontré que les kinases phosphorylent les récepteurs nucléaires et modifient leur activité. Par exemple, la PKA et la MAPK peuvent phosphoryler les récepteurs nucléaires, ce qui démontre la diaphonie entre différentes voies biochimiques (Adams et coll., 1997; Tzagarakis-Foster et Privalsky, 1998). Bien que de nombreux sites différents aient été désignés comme cibles de la phosphorylation dans tous les domaines des récepteurs nucléaires, les résidus de sérine présents dans le domaine A/B sont des cibles communes.
La phosphorylation peut avoir pour effet de favoriser ou de réprimer la transcription. Dans une situation où la phosphorylation permet la liaison des coactivateurs, la transcription serait régulée à la hausse. Cependant, la transcription serait inhibée si la phosphorylation empêche la dimérisation du récepteur (Dongsheng Chen Paul E. Pace et Ali, 1999). Par exemple, la phosphorylation de l’adrénorécepteur par la PKA favorise l’activation dépendante et indépendante du ligand, tandis que la phosphorylation du récepteur alpha des œstrogènes par la PKA peut réduire la dimérisation et la liaison à l’ADN. Toutefois, la phosphorylation de différents sites du récepteur alpha des œstrogènes par d’autres kinases (comme la MAPK) peut favoriser l’activation. Par conséquent, les effets de la phosphorylation de l’activité des récepteurs nucléaires sont complexes et difficiles à prévoir.
Les récepteurs nucléaires se distinguent des autres types de récepteurs par de multiples caractéristiques. Voici quelques-uns des points clés :
Adams, M., Reginato, M. J., Shao, D., Lazar, M. A., et Chatterjee, V. K. (1997). Transcriptional activation by peroxisome proliferator-activated receptor gamma is inhibited by phosphorylation at a consensus mitogen-activated protein kinase site. The Journal of Biological Chemistry, 272(8), 5128-5132. https://www.jbc.org/article/S0021-9258(19)79350-8/fulltext
Dahlman-Wright, K., Koehler, K., et Gustafsson, J.-Å. (2003). Estrogen Receptor-β Structure and Function. In H. L. Henry et A. W. Norman (éd.), Encyclopedia of Hormones (599-608). Academic Press. https://doi.org/10.1016/B0-12-341103-3/00091-7
de Jésus-Tran, K., Côté, P.-L., Cantin, L., Blanchet, J., Labrie, F., et Breton, R. (2006). Comparison of crystal structures of human androgen receptor ligand-binding domain complexed with various agonists reveals molecular determinants responsible for binding affinity. Protein Science, 15(5), 987-999. https://doi.org/10.1110/ps.051905906
Dongsheng Chen Paul E. Pace, R. C. C., et Ali, S. (1999). Phosphorylation of Human Estrogen Receptor α by Protein Kinase A Regulates Dimerization. Molecular and Cellular Biology, 19(2), 1002-1015. https://doi.org/10.1128/MCB.19.2.1002
Feng, Q., et He, B. (2019). Androgen Receptor Signaling in the Development of Castration-Resistant Prostate Cancer. Frontiers in Oncology, 9. https://doi.org/10.3389/fonc.2019.00858
Gomperts, B. D., Kramer, I. M., et Tatham, P. E. R. (2009). Signal transduction . In Signal transduction (2e éd.). Elsevier/Academic Press.
Laudet, V. (1997). Evolution of the nuclear receptor superfamily: early diversification from an ancestral orphan receptor. Journal of Molecular Endocrinology, 19(3), 207-226. https://doi.org/10.1677/jme.0.0190207
Lavery, D. N., et McEwan, I. J. (2005). Structure and function of steroid receptor AF1 transactivation domains: induction of active conformations. In Biochemical journal (vol. 391, no 3). Portland Press Ltd. https://doi.org/10.1042/bj20050872
Li, Y., Lambert, M. H., et Xu, H. E. (2003). Activation of Nuclear Receptors: A Perspective from Structural Genomics. Structure, 11(7), 741-746. https://doi.org/10.1016/S0969-2126(03)00133-3
Mazaira GI, Zgajnar NR, Lotufo CM, Daneri-Becerra C, Sivils JC, Soto OB, Cox MB, G. M. (2018). The Nuclear Receptor Field: A Historical Overview and Future Challenges. Nuclear Receptor Research. https://doi.org/10.11131/2018/101320
Sever, R., et Glass, C. K. (2013). Signaling by nuclear receptors. Cold Spring Harbor Perspectives in Biology, 5(3), a016709-a016709. https://doi.org/10.1101%2Fcshperspect.a016709
Tzagarakis-Foster, C., et Privalsky, M. L. (1998). Phosphorylation of Thyroid Hormone Receptors by Protein Kinase A Regulates DNA Recognition by Specific Inhibition of Receptor Monomer Binding. The Journal of Biological Chemistry, 273(18), 10926-10932. https://doi.org/10.1074/jbc.273.18.10926
Tzavlaki, K., et Moustakas, A. (2020). TGF-β signaling. Biomolecules (Basel, Switzerland), 10(3), 487. https://doi.org/10.3390/biom10030487
Weikum, E. R., Liu, X., et Ortlund, E. A. (2018). The nuclear receptor superfamily: A structural perspective. In Protein Science (vol. 27, no 11, 1876-1892). https://doi.org/10.1002/pro.3496
5
Adams, M., Reginato, M. J., Shao, D., Lazar, M. A., et Chatterjee, V. K. (1997). Transcriptional activation by peroxisome proliferator-activated receptor gamma is inhibited by phosphorylation at a consensus mitogen-activated protein kinase site. The Journal of Biological Chemistry, 272(8), 5128-5132. https://www.jbc.org/article/S0021-9258(19)79350-8/fulltext
Alberts, Bruce. (2002). Molecular biology of the cell. In Molecular biology of the cell (4e éd.). Garland Science. https://www.ncbi.nlm.nih.gov/books/NBK26822/
Bajusz, D., Pándy-Szekeres, G., Takács, Á., de Araujo, E. D., et Keserű, G. M. (2023). SH2db, an information system for the SH2 domain. Nucleic Acids Research, 51(W1), W542-W552. https://doi.org/10.1093/nar/gkad420
Bhagirath, A. Y., Li, Y., Patidar, R., Yerex, K., Ma, X., Kumar, A., et Duan, K. (2019). Two Component Regulatory Systems and Antibiotic Resistance in Gram-Negative Pathogens. International Journal of Molecular Sciences, 20(7). https://doi.org/10.3390/ijms20071781
Bhate, M. P., Molnar, K. S., Goulian, M., et DeGrado, W. F. (2015). Signal Transduction in Histidine Kinases: Insights from New Structures. Structure, 23(6), 981-994. https://doi.org/10.1016/j.str.2015.04.002
Boczek, T., Mackiewicz, J., Sobolczyk, M., Wawrzyniak, J., Lisek, M., Ferenc, B., Guo, F., et Zylinska, L. (2021). The Role of G Protein-Coupled Receptors (GPCRs) and Calcium Signaling in Schizophrenia. Focus on GPCRs Activated by Neurotransmitters and Chemokines. Cells, 10(5). https://doi.org/10.3390/cells10051228
Buday, L., Egan, S. E., Rodriguez Viciana, P., Cantrell, D. A., et Downward, J. (1994). A complex of Grb2 adaptor protein, Sos exchange factor, and a 36-kDa membrane-bound tyrosine phosphoprotein is implicated in ras activation in T cells. Journal of Biological Chemistry, 269(12), 9019-9023. https://doi.org/10.1016/S0021-9258(17)37070-9
Bulavin, D. V, Saito, S., Hollander, M. C., Sakaguchi, K., Anderson, C. W., Appella, E., et Fornace, A. J. (1999). Phosphorylation of human p53 by p38 kinase coordinates N‐terminal phosphorylation and apoptosis in response to UV radiation. The EMBO Journal, 18(23), 6845-6854. https://doi.org/10.1093/emboj/18.23.6845
Buschiazzo, A., et Trajtenberg, F. (2019). Two-Component Sensing and Regulation: How Do Histidine Kinases Talk with Response Regulators at the Molecular Level? Annual Review of Microbiology, 73(1), 507-528. https://doi.org/10.1146/annurev-micro-091018-054627
Calebiro, D., Koszegi, Z., Lanoiselée, Y., Miljus, T., et O’Brien, S. (2021). G protein-coupled receptor-G protein interactions: a single-molecule perspective. Physiological Reviews, 101(3), 857-906. https://doi.org/10.1152/physrev.00021.2020
Dahlman-Wright, K., Koehler, K., et Gustafsson, J.-Å. (2003). Estrogen Receptor-β Structure and Function. In H. L. Henry et A. W. Norman (éd.), Encyclopedia of Hormones (599-608). Academic Press. https://doi.org/10.1016/B0-12-341103-3/00091-7
de Araujo, E. D., Erdogan, F., Neubauer, H. A., Meneksedag-Erol, D., Manaswiyoungkul, P., Eram, M. S., Seo, H. S., Qadree, A. K., Israelian, J., Orlova, A., Suske, T., Pham, H. T. T., Boersma, A., Tangermann, S., Kenner, L., Rülicke, T., Dong, A., Ravichandran, M., Brown, P. J., … Gunning, P. T. (2019). Structural and functional consequences of the STAT5BN642H driver mutation. Nature Communications, 10(1). https://doi.org/10.1038/s41467-019-10422-7
de Araujo, E. D., Keserű, G. M., Gunning, P. T., et Moriggl, R. (2020).Targeting STAT3 and STAT5 in Cancer. Cancers, 12(8). https://doi.org/10.3390/cancers12082002
de Araujo, E. D., Orlova, A., Neubauer, H. A., Bajusz, D., Seo, H.-S., Dhe-Paganon, S., Keserű, G. M., Moriggl, R., et Gunning, P. T. (2019). Structural Implications of STAT3 and STAT5 SH2 Domain Mutations. Cancers, 11(11). https://doi.org/10.3390/cancers11111757
de Jésus-Tran, K., Côté, P.-L., Cantin, L., Blanchet, J., Labrie, F., et Breton, R. (2006). Comparison of crystal structures of human androgen receptor ligand-binding domain complexed with various agonists reveals molecular determinants responsible for binding affinity. Protein Science, 15(5), 987-999. https://doi.org/10.1110/ps.051905906
Dongsheng, C., Pace, P.E., R. C. C., et Ali, S. (1999). Phosphorylation of Human Estrogen Receptor α by Protein Kinase A Regulates Dimerization. Molecular and Cellular Biology, 19(2), 1002-1015. https://doi.org/10.1128/MCB.19.2.1002
Erdogan, F., Radu, T. B., Orlova, A., Qadree, A. K., de Araujo, E. D., Israelian, J., Valent, P., Mustjoki, S. M., Herling, M., Moriggl, R., et Gunning, P. T. (2022). JAK-STAT core cancer pathway: An integrative cancer interactome analysis. Journal of Cellular and Molecular Medicine, 26(7), 2049-2062. https://doi.org/10.1111/jcmm.17228
Feng, Q., et He, B. (2019). Androgen Receptor Signaling in the Development of Castration-Resistant Prostate Cancer. Frontiers in Oncology, 9. https://doi.org/10.3389/fonc.2019.00858
Gaitonde, S. A., et González-Maeso, J. (2017). Contribution of heteromerization to G protein-coupled receptor function. Current Opinion in Pharmacology, 32, 23-31. https://doi.org/10.1016/j.coph.2016.10.006
Glaviano, A., Foo, A. S. C., Lam, H. Y., Yap, K. C. H., Jacot, W., Jones, R. H., Eng, H., Nair, M. G., Makvandi, P., Geoerger, B., Kulke, M. H., Baird, R. D., Prabhu, J. S., Carbone, D., Pecoraro, C., Teh, D. B. L., Sethi, G., Cavalieri, V., Lin, K. H., … Kumar, A. P. (2023). PI3K/AKT/mTOR signaling transduction pathway and targeted therapies in cancer. Molecular Cancer, 22(1), 1-138. https://doi.org/10.1186/s12943-023-01827-6
Gomperts, B. D., Kramer, I. M., et Tatham, P. E. R. (2009). Signal transduction . In Signal transduction (2e éd.). Elsevier/Academic Press.
Gungor, M. Z., Uysal, M., et Senturk, S. (2022). The Bright and the Dark Side of TGF-beta; Signaling in Hepatocellular Carcinoma: Mechanisms, Dysregulation, and Therapeutic Implications. Cancers, 14(4). https://doi.org/10.3390/cancers14040940
Harwood, K. H., McQuade, R. M., Jarnicki, A., et Schneider-Futschik, E. K. (2021). Anti-Inflammatory Influences of Cystic Fibrosis Transmembrane Conductance Regulator Drugs on Lung Inflammation in Cystic Fibrosis. International Journal of Molecular Sciences, 22(14). https://doi.org/10.3390/ijms22147606
Hill, C. S. (2009). Nucleocytoplasmic shuttling of Smad proteins. Cell Research, 19(1), 36-46. https://doi.org/10.1038/cr.2008.325
Ichijo, H., Nishida, E., Irie, K., ten Dijke, P., Saitoh, M., Moriguchi, T., Takagi, M., Matsumoto, K., Miyazono, K., et Gotoh, Y. (1997). Induction of Apoptosis by ASK1, a Mammalian MAPKKK That Activates SAPK/JNK and p38 Signaling Pathways. Science, 275(5296), 90-94. https://doi.org/10.1126/science.275.5296.90
Kadamur, G., et Ross, E. M. (2013). Mammalian phospholipase C. Annual Review of Physiology, 75(1), 127-154. https://doi.org/10.1146/annurev-physiol-030212-183750
Kemp, B. E., Graves, D. J., Benjamini, E., et Krebs, E. G. (1977). Role of multiple basic residues in determining the substrate specificity of cyclic AMP-dependent protein kinase. The Journal of Biological Chemistry, 252(14), 4888-4894. https://www.jbc.org/article/S0021-9258(17)40137-2/pdf
Laudet, V. (1997). Evolution of the nuclear receptor superfamily: early diversification from an ancestral orphan receptor. Journal of Molecular Endocrinology, 19(3), 207-226. https://doi.org/10.1677/jme.0.0190207
Lavery, D. N., et McEwan, I. J. (2005). Structure and function of steroid receptor AF1 transactivation domains: induction of active conformations. In Biochemical journal (vol. 391, no 3). Portland Press Ltd. https://doi.org/10.1042/bj20050872
Lemmon, M. A., et Schlessinger, J. (2010). Cell Signaling by Receptor Tyrosine Kinases. Cell, 141(7), 1117-1134. https://doi.org/10.1016/j.cell.2010.06.011
Li, Y., Lambert, M. H., et Xu, H. E. (2003). Activation of Nuclear Receptors: A Perspective from Structural Genomics. Structure, 11(7), 741-746. https://doi.org/10.1016/S0969-2126(03)00133-3
Liu, Y., et Chance, M. R. (2014). Integrating phosphoproteomics in systems biology. Computational and Structural Biotechnology Journal, 10(17), 90-97. https://doi.org/10.1016%2Fj.csbj.2014.07.003
Maegley, K. A., Admiraal, S. J., et Herschlag, D. (1996). Ras-Catalyzed Hydrolysis of GTP: A New Perspective from Model Studies. Proceedings of the National Academy of Sciences – PNAS, 93(16), 8160-8166. https://doi.org/10.1073/pnas.93.16.8160
Manning, G., Whyte, D. B., Martinez, R., Hunter, T., et Sudarsanam, S. (2002). The Protein Kinase Complement of the Human Genome. Science, 298(5600), 1912-1934. https://doi.org/10.1126/science.1075762
Mazaira GI, Zgajnar NR, Lotufo CM, Daneri-Becerra C, Sivils JC, Soto OB, Cox MB, G. M. (2018). The Nuclear Receptor Field: A Historical Overview and Future Challenges. Nuclear Receptor Research. https://doi.org/10.11131/2018/101320
Meister, M., Tomasovic, A., Banning, A., et Tikkanen, R. (2013). Mitogen-Activated Protein (MAP) Kinase Scaffolding Proteins: A Recount. International Journal of Molecular Sciences, 14(3), 4854-4884. https://doi.org/10.3390/ijms14034854
Montminy, M. R., Gonzalez, G. A., et Yamamoto, K. K. (1990). Regulation of camp-inducible genes by creb. Trends in Neurosciences, 13(5), 184-188. https://doi.org/10.1016/0166-2236(90)90045-C
Moon, S. Y., Kim, K. D., Yoo, J., Lee, J.-H., et Hwangbo, C. (2021). Phytochemicals Targeting JAK–STAT Pathways in Inflammatory Bowel Disease: Insights from Animal Models. Molecules, 26(9). https://doi.org/10.3390/molecules26092824
Morrison, D. K. (2001). KSR: a MAPK scaffold of the Ras pathway? Journal of Cell Science, 114(9), 1609-1612. https://doi.org/10.1242/jcs.114.9.1609
Morrison, D. K. (2012). MAP kinase pathways. Cold Spring Harbor Perspectives in Biology, 4(11), a011254-a011254. https://doi.org/10.1101%2Fcshperspect.a011254.
Osaki, L. H., et Gama, P. (2013). MAPKs and Signal Transduction in the Control of Gastrointestinal Epithelial Cell Proliferation and Differentiation. International Journal of Molecular Sciences, 14(5), 10143-10161. https://doi.org/10.3390/ijms140510143
Papon, N., et Stock, A. M. (2019). Two-component systems. Current Biology, 29(15), R724-R725. https://doi.org/10.1016/j.cub.2019.06.010
Pike, L. J., Eakes, A. T., et Krebs, E. G. (1986). Characterization of affinity-purified insulin receptor/kinase. Effects of dithiothreitol on receptor/kinase function. Journal of Biological Chemistry, 261(8), 3782-3789. https://doi.org/10.1016/S0021-9258(17)35716-2
Qu, C. K. (2000). The SHP—2 tyrosine phosphatase: Signaling mechanisms and biological functions. Cell Research, 10(4), 279-288. https://doi.org/10.1038/sj.cr.7290055
Reich, N. C. (2013). STATs get their move on. JAK-STAT, 2(4), e27080. https://doi.org/10.4161/jkst.27080
Robinson, D. R., Wu, Y.-M., et Lin, S.-F. (2000). The protein tyrosine kinase family of the human genome: Tyrosine Kinases. Oncogene, 19(49), 5548-5557. https://doi.org/10.1038/sj.onc.1203957
S. Corbalan-Garcia S.-S. Yang, K. R. D., et Bar-Sagi, D. (1996).Identification of the Mitogen-Activated Protein Kinase Phosphorylation Sites on Human Sosl That Regulate Interaction with Grb2. Molecular and Cellular Biology, 16(10), 5674-5682. https://doi.org/10.1128/MCB.16.10.5674
Sayem, A. S. M., Arya, A., Karimian, H., Krishnasamy, N., Ashok Hasamnis, A., et Hossain, C. F. (2018). Action of Phytochemicals on Insulin Signaling Pathways Accelerating Glucose Transporter (GLUT4) Protein Translocation. Molecules, 23(2). https://doi.org/10.3390/molecules23020258
Schmid, A. C., Byrne, R. D., Vilar, R., et Woscholski, R. (2004). Bisperoxovanadium compounds are potent PTEN inhibitors. FEBS Letters, 566(1–3), 35-38. https://doi.org/10.1016/j.febslet.2004.03.102
Ségaliny, A. I., Tellez-Gabriel, M., Heymann, M.-F., et Heymann, D. (2015). Receptor tyrosine kinases: Characterisation, mechanism of action and therapeutic interests for bone cancers. Journal of Bone Oncology, 4(1), 1-12. https://doi.org/10.1016/j.jbo.2015.01.001
Seok, S.-H. (2021). Structural Insights into Protein Regulation by Phosphorylation and Substrate Recognition of Protein Kinases/Phosphatases. Life, 11(9). https://doi.org/10.3390/life11090957
Sever, R., et Glass, C. K. (2013). Signaling by nuclear receptors. Cold Spring Harbor Perspectives in Biology, 5(3), a016709-a016709. https://doi.org/10.1101%2Fcshperspect.a016709
Tato, I., Bartrons, R., Ventura, F., et Rosa, J. L. (2011). Amino Acids Activate Mammalian Target of Rapamycin Complex 2 (mTORC2) via PI3K/Akt Signaling. The Journal of Biological Chemistry, 286(8), 6128-6142. https://doi.org/10.1074/jbc.m110.166991
Tzagarakis-Foster, C., et Privalsky, M. L. (1998). Phosphorylation of Thyroid Hormone Receptors by Protein Kinase A Regulates DNA Recognition by Specific Inhibition of Receptor Monomer Binding. The Journal of Biological Chemistry, 273(18), 10926-10932. https://doi.org/10.1074/jbc.273.18.10926
Tzavlaki, K., et Moustakas, A. (2020). TGF-β signaling. Biomolecules (Basel, Switzerland), 10(3), 487. https://doi.org/10.3390/biom10030487
Waksman, G., Shoelson, S. E., Pant, N., Cowburn, D., et Kuriyan, J. (1993). Binding of a high affinity phosphotyrosyl peptide to the Src SH2 domain: Crystal structures of the complexed and peptide-free forms. Cell, 72(5), 779-790. https://doi.org/10.1016/0092-8674(93)90405-F
Weikum, E. R., Liu, X., et Ortlund, E. A. (2018). The nuclear receptor superfamily: A structural perspective. In Protein Science (vol. 27, no 11, 1876-1892). https://doi.org/10.1002/pro.3496
Weis, W. I., et Kobilka, B. K. (2018). The Molecular Basis of G Protein–Coupled Receptor Activation. Annual Review of Biochemistry, 87(1), 897-919. https://doi.org/10.1146/annurev-biochem-060614-033910
Welsh, C. L., Conklin, A. E., et Madan, L. K. (2023). Interaction Networks Explain Holoenzyme Allostery in Protein Kinase A. Kinases and Phosphatases, 1(4), 265-287. https://doi.org/10.3390/kinasesphosphatases1040016
Xiao, Z., Liu, X., Henis, Y. I., et Lodish, H. F. (2000). A Distinct Nuclear Localization Signal in the N Terminus of Smad 3 Determines Its Ligand-Induced Nuclear Translocation. Proceedings of the National Academy of Sciences – PNAS, 97(14), 7853-7858. https://doi.org/10.1073/pnas.97.14.7853
Xu, Y., et Fisher, G. J. (2012). Receptor type protein tyrosine phosphatases (RPTPs) – roles in signal transduction and human disease. Journal of Cell Communication and Signaling, 6(3), 125-138. https://doi.org/10.1007/s12079-012-0171-5
Yoon, M.-S. (2017). The Role of Mammalian Target of Rapamycin (mTOR) in Insulin Signaling. Nutrients, 9(11), 1176. https://doi.org/10.3390/nu9111176
Zheng, C. F., et Guan, K. L. (1993). Properties of MEKs, the kinases that phosphorylate and activate the extracellular signal-regulated kinases. The Journal of Biological Chemistry, 268(32), 23933-23939. https://www.jbc.org/article/S0021-9258(20)80474-8/pdf
Zhou, Y., Prakash, P., Gorfe, A. A., et Hancock, J. F. (2018). Ras and the Plasma Membrane: A Complicated Relationship. Cold Spring Harbor Perspectives in Medicine, 8(10). https://doi.org/10.1101/cshperspect.a031831
1
Image de couverture (PDB ID: 6MBW – Structure du facteur de transcription, Source : De Araujo et coll., Jmol: an open-source Java viewer for chemical structures in 3D, 2019, http://www.jmol.org/).
Fig. 1.1 (Structures des acides aminés, sérine, thréonine et tyrosine. Source : NEUROtiker, Public Domain).
Fig. 1.2 (Figure 1. Source : Seok, 2021, licence CC BY).
Fig. 1.3 (Figure 1. Source : Harwood et coll., 2021, licence CC BY).
Fig. 2.1 (Structure de l’ATP. Source : NEUROtiker, domaine public).
Fig. 2.2 (Molécule du mois : Facteur de croissance épidermique. Source : David S. Goodsell et la RCSB PDB, 2010, licence CC BY 4.0).
Fig. 2.3 (Molécule du mois : Récepteur de l’insuline. Source : David S. Goodsell et la RCSB PDB, 2015, licence CC BY 4.0).
Fig. 2.4 (Molécule du mois : Récepteur de l’insuline. Source : David S. Goodsell et la RCSB PDB, 2015, licence CC BY 4.0).
Fig. 2.5 (Figure 4. Source : Sayem et coll., 2018, licence CC BY 4.0).
Fig. 2.6 (Un interrupteur de lumière blanche sur un mur blanc. Source : Tara Winstead, de Pexels, licence domaine public).
Fig. 2.7 (Figure 1. Source : Meister et coll., 2013, licence CC BY 4.0).
Fig. 2.8 (Figure 1. Source : Osaki et Gama, 2013, licence CC BY 4.0).
Fig. 2.9 (Molécule du mois : Protéines kinases RAF. Source : David S. Goodsell et la RCSB PDB, 2016, licence CC BY 4.0).
Fig. 2.10 (Figure 1. Source : Moon et coll., 2021, licence CC BY 4.0).
Fig. 2.11 (Figure 1. Source : Erdogan et coll., 2022, licence CC BY 4.0).
Fig. 2.12 (Figure 1. Source : Gungor et coll., 2022, licence CC BY 4.0).
Fig. 2.13 (Molécule du mois : Systèmes à deux composants. Source : David S. Goodsell et la RCSB PDB, 2015, licence CC BY 4.0).
Fig. 2.14 (Figure 2. Source : Bhagirath et coll., 2019, licence CC BY 4.0).
Fig. 2.15 (Structure de la GMPc. Source : NEUROtiker, domaine public).
Fig. 3.1 (Molécule du mois : Adrénorécepteurs. Source : David S. Goodsell et la RCSB PDB, 2008, licence CC BY 4.0).
Fig. 3.2 (5′-Guanylyl imidodiphosphate. Source : Yikrazuul, domaine public).
Fig. 3.3 (Molécule du mois : protéines G. Source : David S. Goodsell et la RCSB PDB, 2004, licence CC BY 4.0).
Fig. 3.4 (Figure 2. Source : Seok, 2021, licence CC BY).
Fig. 3.5 (Figure 1. Source : Welsh et coll., 2023, licence CC BY).
Fig. 3.6 (Figure 1. Source : Boczek et coll., 2021, licence CC BY).
Fig. 3.7 (Figure 2. Source : Glaviano et coll., 2023, licence CC BY).
Fig. 4.1 (Structure de la testostérone. Source : NEUROtiker, domaine public).
Fig. 4.2 (Organisation structurelle des récepteurs nucléaires. Source : Boghog2, domaine public).
Fig. 4.3 (Figure 1. Source : Feng et He, 2019, licence CC BY).
2
Cette ressource suit les lignes directrices de Appendix A: Checklist for Accessibility de la ressource Accessibility Toolkit – 2nd Edition.
3
Ce projet a été financé par le gouvernement de l’Ontario.
Les opinions exprimées dans cette publication sont celles de l’auteure et ne reflètent pas nécessairement celles du gouvernement de l’Ontario ou du Consortium ontarien pour l’apprentissage en ligne.
